Mikrobiom
Mikrobiom (původně μικρός (mikrós neboli "malý") a βίος (biós neboli "život" ze starověké řečtiny) je organizované společenství mikroorganismů kolonizující jakýkoliv prostor. Přesněji ji definovali v roce 1988 Whipps et al. jako „mikrobiální společenství zabírající definované stanoviště, které má odlišné fyzikálně-chemické vlastnosti." Termín tedy odkazuje nejen na mikroorganismy přítomné v prostředí, ale také zahrnuje jejich prostředí. V roce 2020 zveřejnil mezinárodní panel odborníků výsledek svých diskusí o definici mikrobiomu. Navrhli definici mikrobiomu založenou na novém uvedení „kompaktního, jasného a komplexního popisu termínu“ původně uvedeného Whipps et al., avšak doplněné o dva hlouběji vysvětlující odstavce. První vysvětlující odstavec se věnuje popisu dynamického charakter mikrobiomu a druhý vysvětlující odstavec jasně odděluje pojem mikrobiota od pojmu mikrobiom.
Mikrobiota je společenství mikroorganizmů skládající se ze všech živých členů tvořících mikrobiom. Většina výzkumníků mikrobiomu souhlasí s tím, že bakterie, archea, houby, řasy a protozoa by měly být považovány za členy mikrobiomu. Kontroverznější je integrace fágů, virů, plazmidů a dalších genetických prvků. Dále Whipps s kolektivem v definici prostředí popsali, ve kterém je mikrobiota přítomna. Zde hraje zásadní roli přítomnost sekundárních metabolitů při zprostředkování složitých mezidruhových interakcí, čímž si mikroorganismy zajišťují přežití v konkurenčním prostředí. Tzv. Quorum sensing neboli komunikace mezi mikroorganismy je indukovaná malými molekulami, které mikroorganismy produkují. Toto umožňuje bakteriím řídit kooperativní aktivity a přizpůsobovat jejich fenotypy biotickému prostředí, což vede např. k mezibuněčným adhezím nebo tvorbě biofilmu.
Všechna zvířata a rostliny vytváří vzájemné asociace s různými mikroorganismy, včetně protozoí, bakterií, archeí, hub a virů. V oceánu byly tyto vztahy historicky v systémech jednoho hostitele a jednoho symbionta. Nové výzkumy studující diverzitu mikroorganismů asociovaných s různými hostiteli mořských živočichů však posouvají pole zájmu do studií, které se zabývají interakcemi mezi hostitelem a vícečlenným mikrobiomem. Potenciál mikrobiomu ovlivňovat zdraví, fyziologii, chování a ekologii mořských živočichů by mohl především změnit současné chápání toho, jak se mořští živočichové přizpůsobují změnám. To se týká zejména rostoucích změn souvisejících s klimatem a lidskou činností, které dnes výrazně ovlivňují život v oceánu. Mikrobiom rostlin hraje klíčovou roli pro růst rostlin a následnou produkci potravin z nich. V posledních letech mu proto byla věnována zvýšená pozornost. Rostliny žijí v úzkém spojení s různými mikrobiálními společenstvi, označovanými jako rostlinný mikrobiom žijícími jak uvnitř (endosféra), tak vně (episféra) rostlinných tkání. Rostlinný mikrobiom hraje stěžejní roli v ekologii a fyziologii rostlin. Předpokládá se, že základní rostlinný mikrobiom obsahuje mikrobiální druhy nezbytné pro zdraví rostlin a pro schopnost růstu rostliny. Podobně jako u rostlin se savčí střevní mikrobiom ukázal jako klíčový regulátor fyziologie hostitele. Následná koevoluce mezi hostitelem a jeho střevní mikrobiotou hraje klíčovou roli u adaptace savců na jejich rozmanitý životní styl.
Zájem o výzkum mikrobiomu se začal objevovat v mikrobiologii již v sedmnáctém století. Vývoj nových technik a výrazný posun v laboratorním vybavení podpořil mikrobiologický výzkum, který zásadním způsobem změnil chápání problematiky zdraví a nemocí. Vývoj prvních mikroskopů umožnil objev nového, doposud neznámého a neviditelného světa a vedl k identifikaci jednotlivých mikroorganismů. Infekční nemoci se staly hlavním pilířem zájmu a výzkumu, ačkoliv dnes víme, že pouze malá část mikroorganismů je spojena s vyvoláním onemocnění, a tedy patogenitou. Převážná většina mikrobů je nezbytná pro zdravé fungování těla či jakékoliv jiného ekosystému a je známá prospěšnými interakcemi s jinými mikroorganismy. Koncept existence mikroorganismu jako jednotlivé buňky se brzy začal měnit, a to tehdy, když bylo stále zjevnější, že mikroorganismy se vyskytují v komplexních sdruženích (konsorciích), ve kterých jsou kritické interakce druhů a vzájemné mezidruhové komunikace. Objev DNA, vývoj sekvenačních technologií, PCR a klonovacích technik umožnily podrobnější zkoumání mikrobiálních komunit pomocí přístupů nezávislých na jejich kultivaci. K dalším změnám došlo na začátku tohoto století a stále pokračují, protože nové technologie sekvenování a nashromážděná sekvenční data zdůraznily jednak všudypřítomnost mikrobiálních komunit ve spojení s vyššími organismy, ale také kritickou roli mikrobů ve zdraví lidí, zvířat a rostlin. Tato zjištění způsobila revoluci v mikrobiální ekologii. Analýza genomů a metagenomů vysoce výkonným způsobem nyní poskytuje vysoce efektivní metody pro výzkum fungování jednotlivých mikroorganismů i celých mikrobiálních společenstev v jednotlivých přírodních stanovištích či hostujících organismech.
Základní informace
Historie
Zájem o výzkum mikrobiomu se v mikrobiologii začal objevovat již v sedmnáctém století. Vývoj nových technik a laboratorních zařízení zintenzivnili mikrobiologický výzkum a způsobili změny v porozumění kontextu stavu zdraví a nemocí. Vzhledem k tomu, že infekční choroby postihovaly lidskou populaci již od dávné minulosti, byla lékařská mikrobiologie prvním cílem výzkumu a veřejného zájmu. Vývoj prvních mikroskopů umožnil objev nového, neznámého světa a vedl k identifikaci mikroorganismů.
- Změna paradigmatu
- Posun paradigmatu směrem od mikrobů jakožto samostatných organismů způsobujících nemoci k holistickému pohledu na mikroorganismy jako na komunitu propojující všechny oblasti lidského života.[1]
Přístup do dříve neviditelného světa otevřel oči a mysl badatelům sedmnáctého století. Antonie van Leeuwenhoek zkoumal rozmanité bakterie různých tvarů, houby a prvoky, které nazval animalcules, a to především ze vzorků vody, bahna a zubního plaku. Zároveň objevil biofilmy jako první známku interakcí mikroorganismů v rámci komplexních komunit. Významným mezníkem v mikrobiologii bylo převratné zjištění Roberta Kocha, které vysvětlovalo původ nemocí lidí a zvířat jako důsledek mikrobiální infekce, a také vysvětlovalo koncept patogenity. Tato zjištění posunula zaměření vědců a veřejnosti na roli mikroorganismů jako choroboplodných činitelů, které je třeba eliminovat.
Komplexní výzkum vedený v minulém století však ukázal, že pouze malá část mikroorganismů je spojena s onemocněním nebo patogenitou. Převážná většina mikrobů je nezbytná pro fungování ekosystému a je známá prospěšnými interakcemi s jinými mikroby, stejně tak jako s makroorganismy. Ve skutečnosti je udržování zdravého mikrobiomu pro lidské zdraví zásadní a může být potenciálním cílem pro nová terapeutika.[2] Na konci devatenáctého století byla zahájena éra mikrobiální ekologie průkopnickým dílem Martinuse W. Beijerincka a Sergeje Winogradského. Nově založená věda o mikrobiologii životního prostředí vedla k další změně paradigmatu: mikroorganismy jsou všude v přirozeném prostředí, často spojené s hostiteli, přičemž na hostitele mají příznivé účinky. Toto tvrzení zde bylo uvedeno vůbec poprvé.[3][4]
Následně se koncept, že se mikroorganismy v prostředí chovají jako samostatné, jednotlivé buňky, začal měnit. Důvodem byl stále zjevnější fakt, že se mikroorganismy vyskytují v komplexních sdruženích, ve kterých jsou interakce jednotlivých druhů a jejich komunikace rozhodující pro dynamiku celé populace a také pro funkční aktivitu celého uskupení.[5] Objev DNA, vývoj sekvenačních technologií, PCR a klonovacích technik umožnily zkoumání mikrobiálních komunit pomocí přístupů nezávislých na kultivaci, založených na DNA a RNA.[6]
Dalším milníkem bylo zavedení fylogenetických markerů. Tím hlavním se stal gen pro 16S rRNA podjednotku, pomocí kterého bylo možné taxonomicky určit jednotlivé druhy. O tento posun v analýze mikrobiálních komunit se zasloužili Carl Woese a George E. Fox v roce 1977.[7] V dnešní době mohou biologové sledovat přítomnost bakterií, archeí, hub, řas a protozoí v jejich přirozeném prostředí, např. zacílením na jejich geny 16S a 18S rRNA, vnitřní transkribovaný spacer (ITS) u hub a mikromycet, anebo alternativně na specifické funkční oblasti genů kódujících specifické enzymy.[8][9][10]
Další velká změna ve vnímání problematiky studia mikrobiomu byla zahájena na začátku tohoto století a pokračuje až do současnosti, protože nové sekvenační technologie a stále vznikající sekvenční data zdůraznily všudypřítomnost mikrobiálních komunit ve spojení s vyššími organismy a kritickou roli mikrobů pro zdraví lidí, zvířat i rostlin.[11] Nové sekvenační možnosti způsobily revoluci v mikrobiální ekologii, protože analýza genomů a metagenomů vysoce výkonným způsobem poskytuje účinnou a efektivní metodu pro objevování funkčního potenciálu jednotlivých mikroorganismů i celých komunit v jejich přirozeném prostředí.[12][13] Multi-omické technologie včetně metatranskriptomiky, metaproteomiky a metabolomiky nyní poskytují podrobné informace o mikrobiálních aktivitách v prostředí, které kolonizují. Na základě bohaté databáze dat získala kultivace mikrobů, která byla v posledních třiceti letech často podceňována, nový význam a vysoce výkonná kulturomika je nyní důležitým nástrojem pro studium mikrobiomů. Vysoký potenciál a síla kombinace více „omických“ technik pro analýzu interakcí hostitel-mikrob je zdůrazněna v několika publikacích.[14][15]
| Časová osa studia mikrobiomu od 17. století do současnosti | ||||
|---|---|---|---|---|
Úroveň technického pokroku | Rok | vědecké objevy | Vědec | Zdroj |
| mikroskopie | 1670 | objevení mikroorganismů | Antonie van Leeuwenhoek otec mikrobiologie | [16] |
| 1729 | klasifikace rostlin a hub | Pier Antonio Micheli | ||
| 1796 | první vakcinace | Edward Jenner | [17] | |
| 1837 | kvasinky a alkoholová fermentace | Charles de la Tour Friedrich Kützing Theodor Schwann | ||
| kultivační postupy | 1855 -1857 | pasterizace, fermentace, vakcína proti vzteklině | Louis Pasteur | [18] |
| 1875 | základ pro bakteriální taxonomii | Ferdinand Cohn | ||
| 1884 | Kochovy postuláty | Robert Koch | ||
| 1888 | začátek mikrobiální ekologie nitrifikace, fixace dusíku, | Sergei Winogradsky | [19] | |
| 1892 | extrakce viru tabákové mozaiky z listů | Dmitrij Ivanovskij Martinus Beijerinck | ||
| 1904 | koncept rhizosféry | Lorenz Hiltner | ||
| fluorescenční mikroskopie | 1911 | [20] | ||
| hmotnostní spektrometrie | 1919 | Francis Aston | ||
| 1922 | chemolitotrofie | Sergei Winogradsky | [21] | |
| 1928 | transformace genetické informace na potomstvo | Frederick Griffith | [22] | |
| 1928 | objevení antibiotik | Alexander Fleming | ||
| Rastrovací elektronová mikroskopie | 1931 -1938 | |||
| 1944 | DNA jakožto nositelka genetické informace | Oswald Avery Colin Macleod Maclyn McCarty | [23] | |
| 1946 | "sexuální reprodukce" u bakterií | Joshua Lederberg Edward Tatum | [24] | |
| 1953 | 3D struktura dvojité šroubovice[25] | James Watson Francis Crick | ||
| in situ hybridizace iSIS | 1969 | [26] | ||
| HPLC | 1970s | centrální dogma molekulární biologie[27] | Francis Crick | [28] |
| DNA čipy/hybridizace kolonií | 1975 | [29] | ||
| Sangerova metoda sekvenování | 1977 | Frederick Sanger | [30][31] | |
| 1977 | obejvení domény Archaea | Carl Woese George E. Fox | [7][32] | |
| 1977 | první kompletně sekvenovaný genom viru | [33] | ||
| 1982 | objev prionů | Stanley B. Prusiner | [34] | |
| Technologie PCR | 1983 | [35] | ||
| fluorescence in situ hybridisation | 1988 | [36][37] | ||
| 1991 | terorie holobionta | Eugene Rosenberg Ilana Zilber-Rosenberg | [38] | |
| kvalitativní real-time PCR | 1993 | [39] | ||
| 1993 | komplexní struktura biofilmu | Hans-Curt Flemming | [40] | |
| přístup zcela založený na rRNA | 1995 | [41] | ||
| 1995 | první sekvenace celého genomu Haemophilus influenzae | Craig Venter a kolektiv | [42] | |
| Sekvenování nové generace | 2005 | [43] | ||
| 2005 | HMP: Human Microbiome Project | [44] | ||
| Sekvenování třetí generace | 2008 | [45] | ||
| 2008 | TerraGenome: Reference Soil Metagenome Project | [37] | ||
| 2010 | Projekt mikrobiomu na Zemi | [46] | ||
Etymologie
Slovo mikrobiom (z řeckého micro - „malý“ a bíos - „život“) poprvé použil J.L. Mohr v roce 1952 v časopisu The Scientific Monthly pro označení mikroorganismů, které se nacházejících ve specifickém, definovaném prostředí.
Definice
Mikrobiální společenství byla dříve běžně definována jednoduše jako uskupení mikroorganismů žijících společně. Pro upřesnění je vhodnější tvrzení, že mikrobiální společenstva jsou definována jako vícedruhová uskupení, ve kterých mikroorganismy a také další vyšší organismy na sebe vzájemně působí v definovaném prostředí.[47] V roce 1988 Whipps et al. poskytli ve své práci zaměřená na rhizobiální mikroorganismy první definici termínu mikrobiom.[48] Pomocí kombinace slov mikro a biom popsali „charakteristickou mikrobiální komunitu“ v „dobře definovaném prostředí, které má odlišné fyzikálně-chemické vlastnosti“. Tato definice představuje podstatný pokrok v definici mikrobiálního společenstva, protože definuje mikrobiální společenstvo s odlišnými vlastnostmi a funkcemi. Zavádí se nově také popis jeho interakcí s okolním prostředím, což vede k vytvoření specifických ekologických nik.
Za posledních několik desetiletích však bylo publikováno mnoho dalších definic mikrobiomu. Do roku 2020 byla nejcitovanější definice od Lederberg[49], která popsala mikrobiom v ekologickém kontextu jako společenství komenzálních, symbiotických a patogenních mikroorganismů v těle hostitele či v jiném prostředí. Další definice od Marchesi a Ravel se zaměřuje na genomy a popis mikrobiální (a také virové) genové exprese, jejich proteomů v definovaném prostředí.[50] Všechny tyto definice naznačují, že obecné koncepty makroekologie lze snadno aplikovat na ekologii na mezimikrobiální úrovni, stejně jako na interakce mikrob-hostitel. Do jaké míry však lze tyto koncepty vyvinuté pro eukaryota aplikovat na prokaryota s jejich odlišným životním cyklem, pokud jde o dormanci, variace fenotypu a horizontální přenos genů[51] není dosud zcela jasné. Tento fakt s sebou přináší otázku, zda by bylo řešením pro studium mikrobiomu uvažovat zcela nový soubor koncepčních ekologických modelů a teorií pro ekologii mikrobiomů, a to zejména ve vztahu k různým hierarchiím interakcí mikrobů mezi sebou. Mnoho současných definic nedokáže zachytit tuto složitost a popisuje termín mikrobiom tak, že zahrnuje pouze genomy mikroorganismů.
| Definice mikrobiomu | |
|---|---|
| Typ definice | Příklady |
| Ekologický | Definice založené na ekologii popisují mikrobiom podle konceptů odvozených z ekologie mnohobuněčných organismů. Hlavním problémem zde je, že teorie z makroekologie ne vždy odpovídají pravidlům v mikrobiálním světě. |
| |
| Závislé na organismech/hostiteli | Definice závislé na hostiteli jsou založeny na mikrobiálních interakcích mikroorganismů s hostitelem. Hlavní mezery v těchto definicích se týkají otázky, zda lze data o interakcích v jednom hostiteli přenést na jiného hostitele. Pochopení koevoluce a výběru v definicích závislých na hostiteli je také nedostatečně zastoupeno. |
| |
| Genomický / řízený metodou | K dispozici je celá řada definic mikrobiomu, které jsou řízeny použitými metodami. Tyto definice se většinou opírají o analýzu založenou na sekvenci DNA a popisují mikrobiom jako kolektivní genom mikroorganismů v definovaném prostředí. Hlavním nedostatkem je, že každá nová dostupná technologie bude mít za následek potřebu nové definice. |
| |
| Kombinovaný | K dispozici jsou některé definice mikrobiomu, které svými výhodami a nevýhodami vyhovují několika kategoriím. |
| |
V roce 2020 panel odborníků organizovaný projektem MicrobiomeSupport, který byl financovaným EU[62], zveřejnil výsledky svých úvah o definici mikrobiomu. Panel se skládal ze zhruba 40 lídrů z různých oblastí výzkumu mikrobiomů a na sto dalších odborníků z celého světa navíc přispělo prostřednictvím online průzkumu. Toto uskupemí navrhlo definici mikrobiomu založenou především na vyzdvižení toho, co charakterizovali jako „kompaktní, jasný a komplexní popis termínu“ v původní publikaci autoři Whipps et al. v roce 1988.[48] Toto dále doplnili souborem doporučení s ohledem na následný technologický vývoj a výsledky výzkumu. Jasně oddělili pojmy mikrobiom a mikrobiota a poskytli komplexní diskusi s ohledem na složení mikrobiomu, jeho heterogenitu a dynamiku v čase a prostoru, stabilitu a odolnost mikrobiálních komunit, definici základního mikrobiomu a jeho klíčové druhy, dále pak také specifikovali koevoluční principy interakcí mikrob-hostitel a mezidruhové interakce v rámci mikrobiomu.[1]
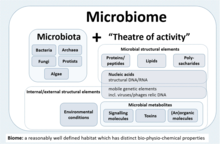
Tento panel rozšířil definici podanou autory Whipps et al., která obsahuje všechny důležité body platné i 30 let po jejím zveřejnění v roce 1988, dvěma vysvětlujícími odstavci odlišujícími pojmy mikrobiom a mikrobiota a vyjadřujícími její dynamický charakter takto:
- Mikrobiom je definován jako charakteristická mikrobiální komunita zabírající dobře definované stanoviště, které má odlišné fyzikálně-chemické vlastnosti. Mikrobiom se nevztahuje pouze na zúčastněné mikroorganismy, ale také zahrnuje jejich dějiště aktivity, což vede k vytvoření specifických ekologických nik. Mikrobiom, který tvoří dynamický a interaktivní mikroekosystém náchylný ke změnám v čase a měřítku, je integrován do makroekosystémů včetně eukaryotických hostitelů, a zde hraje zásadní roli pro jejich fungování a zdraví.
- Mikrobiota se skládá z uskupení mikroorganismů patřících do různých říší (prokaryota-bakterie, archaea; eukaryota-řasy, prvoci, houby atd.), zatímco „jejich dějiště aktivity“ zahrnuje mikrobiální struktury, metabolity, mobilní genetické prvky (jako jsou transpozony), fágy a viry) a reliktní DNA zasazená do podmínek prostředí daného stanoviště.
Členové
Mikrobiota
Mikrobiota zahrnuje všechny živé organismy, které jsou součástí mikrobiomu. Většina vědců studující mikrobiom souhlasí s tím, že bakterie, archaea, houby, řasy a protozoa by měla být považována za členy mikrobiomu.[50] Integrace fágů, virů, plazmidů a mobilních genetických elementů je potom už poněkud kontroverznější otázkou v definici mikrobiomu. Jednoznačná shoda neexistuje ani v tom, zda extracelulární DNA pocházející z mrtvých buněk, tzv. „reliktní DNA“, má být zahrnuta do mikrobiomu nebo ne.[63][1] Reliktní DNA může tvořit až 40 % sekvenované DNA v půdě[64] a při širší analýze biotopů tvoří v průměru až 33 % celkové bakteriální DNA s nejvyšším podílem 80 % v některých vzorcích.[65] Navzdory své všudypřítomnosti měla reliktní DNA minimální vliv na určení taxonomické a fylogenetické diverzity.[65][1]
Pokud jde o použití konkrétních termínů, jasné rozlišení mezi pojmy "mikrobiom" a "mikrobiota" pomáhá vyhnout se kontroverzi týkající se členů zahrnutých v jednotlivých pojmech. Mikrobiota je obvykle definována jako shromáždění živých mikroorganismů přítomných v definovaném prostředí.[50] Fágy, viry, plazmidy, priony, viroidy a volná reliktní DNA nejsou obvykle považovány za živé mikroorganismy,[66] a tedy tím pádem nepatří do mikrobioty a nejsou v této definici zahrnuty.[1]
Jak již bylo několikrát zmíněno, termín mikrobiom, tak jak byl původně postulován v práci od Whipps et al.,[48] zahrnuje nejen společenství mikroorganismů, ale také jejich „dějiště aktivity“. V něm je zahrnuto celé spektrum molekul produkovaných mikroorganismy, včetně jejich strukturních jednotek (nukleové kyseliny, proteiny, lipidy, polysacharidy), metabolitů (signální molekuly, toxiny, organické a anorganické molekuly) a molekul produkovaných koexistujícími hostiteli a strukturované podle okolních podmínek prostředí. Proto by všechny mobilní genetické elementy, jako jsou fágy, viry a „reliktní“ a extracelulární DNA, měly být zahrnuty do termínu mikrobiom, ale nejsou součástí mikrobioty. Termín mikrobiom je také někdy zaměňován s metagenomem. Metagenom je však jasně definován jako soubor genomů a genů členů mikrobioty.[50]
Studie mikrobiomu se někdy zaměřují na chování konkrétní skupiny mikrobioty. Ve vědecké literatuře se začalo objevovat stále více termínů jako bakteriom, archeom, mykobiom nebo virom, ale tyto termíny se nevztahují na biomy, ve stejném slova smyslu jako samotný mikrobiom. V důsledku toho by bylo lepší používat původní termíny, tedy bakteriální, archeální nebo houbové společenství. Na rozdíl od mikrobioty, kterou lze studovat samostatně, je mikrobiom vždy složen ze všech členů, kteří se vzájemně ovlivňují, žijí ve stejných podmínkách a prostředí a společně tvoří svou vlastní ekologickou niku. Dobře zavedený termín virom je odvozen od viru a genomu a používá se k popisu virových metagenomů.[67] Virové metagenomy lze navrhnout jako vědecky lepší a přesnější termín.[1]
Sítě
Mikroorganismy se vzájemně ovlivňují a jejich symbiotické interakce mají různé důsledky na stav mikrobiální populace, populační dynamiku a funkční zastoupení v rámci mikrobiomu.[68] Mikrobiální interakce mohou být buď mezi mikroorganismy stejného druhu nebo mezi různými druhy, rody, rodinami a doménami. Interakce mezi mikroorganismy lze rozdělit na pozitivní, negativní a neutrální. Pozitivní interakce zahrnují mutualismus, synergismus a komenzálismus. Negativní interakce zahrnují amensalismus, jako je predace, parazitismus, antagonismus a konkurence. Neutrální interakce jsou interakce, kde není pozorován žádný vliv na funkčnost nebo vitalitu interagujících mikrobiální druhů.
- Sítě společného výskytu ukazují rozdíl ve střevní mikrobiotě mezi býložravými a masožravými cichlidami
Uzly (kolečka) jsou zbarvené podle kmene. Síť býložravců (Herbivores) má vyšší komplexitu (156 uzlů a 339 propojení) ve srovnání se sítí masožravců (Carnivores) (21 uzlů a 70 propojení).[69]
Různé mikrobiomy vykazují různé adaptivní strategie. Oligotrofové jsou organismy, které zvládají žít v prostředí s velmi nízkými hladinami živin, zejména uhlíku. Vyznačují se zpomaleným růstem, nízkou rychlostí metabolismu a obecně nízkou hustotou populace. Oligotrofní prostředí zahrnuje typicky oceánské sedimenty, jeskyně, ledovcový a polární led, hlubokou půdu, oceánské vody a vyluhované půdy. V kontrastu s nimi jsou kopiotrofy, kterým se daří růst v mnohem vyšších koncentracích uhlíku a dobře se jim daří v podmínkách s vysokým obsahem organického substrátu, jako jsou místa, kde se shromažďují odpadní vody.[70][71]
Koevoluce
Podle „separačního“ přístupu lze mikroorganismy rozdělit na patogeny, neutrální a symbionty podle jejich interakcí s hostitelským organismem. Koevoluce mezi hostitelem a s ním spojenou mikrobiotou může být popsána jako antagonistická (založená na negativních vzájemných interakcích) nebo mutualistická (založená na pozitivních vzájemných interakcích).[72]
Od roku 2020 se začal objevovat zvýšený počet publikací o oportunních patogenech a patobiontech, který způsobil posun v myšlení směrem k holistickému přístupu v koevoluční teorii. Holistický přístup popisuje hostitele a jeho mikrobiotu jako jednu jednotku (takzvaný holobiont ), která se společně vyvíjí a mění jako jedna entita. Podle holistického přístupu je patologický stav holobionta spojen s dysbiózou, nízkou diverzitou přidružené mikrobioty a jejich nízkou variabilitou: nastává tzv. patobiomový stav. Zdravý stav je na druhé straně doprovázen eubiózou, vysokou diverzitou a jednotnost příslušné mikrobioty.
Typy mikrobiomů
Mořský
- Vztah hostitel-mikrobiom mořských živočichů
- Obecně se předpokládá, že vztahy v rámci mořského mikrobiomu jsou v symbiotickém stavu a jsou normálně vystaveny environmentálním a zvířecím specifickým faktorům způsobující přirozené variace. Některé události mohou změnit vztah ve stále fungující, ale již změněný symbiotický stav, zatímco extrémní stresové události mohou způsobit dysbiózu nebo rozpad vztahu a interakcí.[73]
Všichni živočichové na Zemi vytváří asociace s mikroorganismy, včetně protozoí, bakterií, archeí, hub a virů. V oceánu byly historicky zkoumány vztahy mezi živočichy a mikroorganismy v systémech jediného hostitele a symbionta. Nové výzkumy rozmanitosti mikroorganismů asociovaných s různými hostiteli mořských živočichů však posouvají veškerý zájem směrem do studií, které se zabývají interakcemi mezi hostitelem a vícečlenným mikrobiomem. Potenciál mikrobiomu ovlivňovat zdraví, fyziologii, chování a ekologii mořských živočichů by mohl změnit současné vnímání toho, jak se mořští živočichové přizpůsobují změnám, v současné zejména rostoucím klimatickým změnám a změnám způsobeným lidskou činností.
V současné době se studují mikrobiomy různých mořských živočichů, od zjednodušených organismů jako jsou mořské houby[74] a ctenofory[75] až po složitější a komplexnější organismy, jako jsou sumky[76] a žraloci.[77]
Vztah mezi Havajskou olihní a bioluminiscenční bakterií Aliivibrio fischeri je jedním z nejlépe prostudovaných symbiotických vztahů hostitele a mikroorganismu v mořském prostředí a je to systém vhodný pro obecný výzkum symbiózy. Tento vzájemný vztah poskytl vhled do základních procesů ve živočišných a mikrobiálních symbiózách a zejména biochemických interakcí a molekulární signalizace mezi hostitelem a bakterií.[78][79]
Dalším dobře prozkoumaným mořským hostitelem je mořský červ Olavius algarvensis. Tito zhruba tři centimetry dlouzí červi osidlují mělké mořské sedimenty Středozemního moře. Tyto organismy nemají ústa ani trávicí či vylučovací systém, ale místo toho jsou vyživováni pomocí extracelulárních bakteriálních endosymbiontů, kteří jsou schopni koordinovaně využívat síru přítomnou v prostředí.[80] Zmíněný systém studia symbiózy dokáže využívají sofistikovaní "omické metody" a vizualizační nástroje. Například víceznačkové sondování pomocí prób zlepšilo vizualizaci mikrobiomu.[81] Omické metody jako transkriptomika a proteomika byly použity ke zkoumání interakcí hostitel-mikrobiom, včetně studia přenosu energie mezi hostitelem a mikroorganismy[82] a rozpoznání konsorcií pomocí imunitního systému červa.[83] Hlavní předností tohoto systému host-mikrobiom je, že umožňuje studovat interakce s mikrobiálním konsorciem i s nízkou diverzitou a také nabízí řadu hostitelských a mikrobiálních genomických zdrojů[84][85]

Korály jsou jedním z běžných příkladů hostitele, jehož symbióza s mikrořasami se může změnit v dysbiózu a je viditelná jako vybělování korálů. Korálové mikrobiomy byly zkoumány v různých studiích, které demonstrují, jak změny odehrvající se v oceánském prostředí, zejména změna teploty, světla a složení anorganických živin, ovlivňují množství a výkonnost symbiontů mikrořas, a jak tyto změny přispívají k jevům jako kalcifikace a kompletní změna fyziologii hostitele.[87][88] Studie také naznačují, že přítomné bakterie, archea a houby navíc přispívají k koloběhu živin a organické hmoty v korálech, přičemž viry mohou také hrát roli při ustalování složení. Tyto studie tak poskytují jeden z prvních pohledů na multi-doménovou symbiózu mořských živočichů.[89] Gamaproteobakterie Endozoicomonas se objevuje jako stěžejní člen mikrobiomu korálů s vysokou flexibilitou potřebných životních podmínek.[86][90] Vzhledem k nedávnému masovému vybělování korálů, které se odehrává na útesech[91] budou korály pravděpodobně i nadále užitečným a důležitým systémem pro výzkum symbiózy a dysbiózy.
Houbovci jsou běžnými členy různých biotopů oceánu a jejich četnost v oceánském prostředí a schopnost filtrovat velké objemy mořské vody vedou k domněnce, že tyto organismy hrají zásadní roli při ovlivňování procesů odehrávajících se v bentických a pelagických místech v oceánu.[92] Jsou jednou z nejstarších eukaryot a mají relativně jednoduchou stavbu těla, který se běžně spojuje s bakteriemi, archeami, prvoky, houbami a viry.[93] Studie dokazují, že mikrobiom houbovců přispívá k koloběhu dusíku v oceánech, zejména prostřednictvím oxidace amoniaku.[94][95] Nedávno bylo prokázáno, že mikrobiální symbionti tropických hub produkují a uchovávají polyfosfátová granula[96], což pravděpodobně umožňuje hostiteli přežít období nedostatku fosfátů v oligotrofních mořských prostředích a jejich vyčerpání.[97] Zdá se, že mikrobiomy některých druhů hub mění své složení v reakci na měnící se podmínky prostředí, včetně teploty[98] a okyselení oceánů.[99][100]
- Odběr vzorku z proudu vody tryskajícího po vynoření modré velryby pomocí dronu vrtulníku[101]
- Relativní množství bakteriálních tříd ze vzorků velryb, vzduchu a mořské vody.[102]
Mikrobiom kytovců je obtížné studovat kvůli potížím s dostupností a přístupem k odebíraným vzorkům. Mnoho druhů velryb patří mezi vzácné druhy a navíc se potápí do hlubin oceánu. Existují různé techniky pro odběr vzorků střevního mikrobiomu kytovců. Nejběžnější je odběr vzorků stolice z prostředí a následně odběr sondou z místa, které nebylo kontaminované.[103] Kůže je bariéra chránící mořské savce před vnějším světem. Epidermální mikrobiom, který se vyskytuje na kůži živočichů, je indikátorem nejenom zdravotního stavu zvířete, ale je také ekologickým indikátorem stavu okolního prostředí. Znalost toho, jak vypadá mikrobiom kůže mořských savců v jejich běžných životních podmínkách nám umožňuje pochopit, jak se tato společenství liší od volných mikrobiálních společenství nalezených v moři.[104] Kytovci jsou v ohrožení především proto, že jsou vystaveni mnoha stresovým faktorům, které je činí zranitelnějším a náchylnějším vůči různým nemocem. Je známá vysoká náchylnost kytovců k infekcím dýchacích cest, ale o jejich respiračním mikrobiomu je známo jen málo. Odběr vzorků vydechovaného dechu neboli „foukání“ kytovců může poskytnout dobrý materiál k posouzení jejich zdravotního stavu. Tato vyfukovaná kapalina se skládá ze směsi mikroorganismů a organického materiálu, včetně lipidů, proteinů a buněčných zbytků z výstelky dýchacích cest, které po uvolnění do relativně chladnějšího venkovního vzduchu kondenzují a vytvářejí viditelnou masu páry, která je při vzorkování odebírána. Existují různé metody pro sběr vzorků vydechovaného kondenzovaného plynu. V současnosti se používá především jedna z nejnovějších, a to pomocí leteckých dronů. Tato metoda poskytuje bezpečnější, tišší a méně invazivní alternativu a často cenově výhodnou možnost monitorování a odběru vzorku. Vzorky vyfoukované páry kondenzující v kapalinu se odebírají do laboratoře, kde se dále zpracovávají a dochází k amplifikaci a sekvenaci DNA pocházející z mikrobioty kolonizující dýchací cesty kytovce. Použití vzdušných dronů je obecně úspěšnější u velkých kytovců, a to kvůli pomalé rychlosti plavání a většího objemu vyfukovaného plynu.[105][106][107]
Pozemní mikrobiom
Rostliny
- Mikrobiom v rostlinném ekosystému
Rostlinný mikrobiom hraje výraznou roli ve schopnosti zdravého růstu rostlin, s čímž je úzce spojena dostatečná produkce potravin vyráběných z rostlin. Z tohoto důvodu mu v posledních letech byla věnována značná pozornost.[108] Rostliny žijí v úzkém spojení s různými mikrobiálními konsorcii. Tyto mikroorganismy, označované jako rostlinná mikrobiota, žijí jak uvnitř (endosféra), tak i vně (episféra) rostlinných tkání a hrají důležitou roli v ekologii a fyziologii rostlin. "Předpokládá se, že základní rostlinná mikrobiota obsahuje takové taxony, které jsou důležité pro dobrý růst rostlin a jejichž přítomnost byla dlouhodobě definována prostřednictvím evolučních mechanismů selekce, čímž došlo k obohacení mikrobiálních taxonů obsahujících geny základních funkcí pro celkový prospěšný růst rostlinného holobionta."[109]
Rostlinné mikrobiomy jsou utvářeny jednak faktory souvisejícími s rostlinou samotnou, jako je její genotyp, druh a rostlinný orgán, kde ke kolonizaci dochází, tak i faktory souvisejícími s prostředím rostliny, jako je okolní hospodaření, využívání půdy a místní klima.[110] V některých studiích se uvádí, že důležitý je rovněž zdravotní stav rostliny, který odráží její mikrobiom a úzce s ním souvisí.[111][108][112]
Rostlinná mikrobiota kolonizuje různé niky vně i uvnitř rostlinné tkáně. Všechny nadzemní části rostlin dohromady, nazývané fylosféra, se neustále vyvíjejí v důsledku expozice ultrafialového (UV) záření a měnících se klimatických podmínek. Skládá se především z listů. Podzemní části rostlin, především kořeny, jsou obecně ovlivňovány vlastnostmi půdy. Škodlivé interakce některých členů mikrobioty vlivem své patogenní aktivity negativně ovlivňují růst rostlin. Na druhou stranu, prospěšné vzájemné mikrobiální interakce v podzemní části podporují růst rostlin.
Zvířata
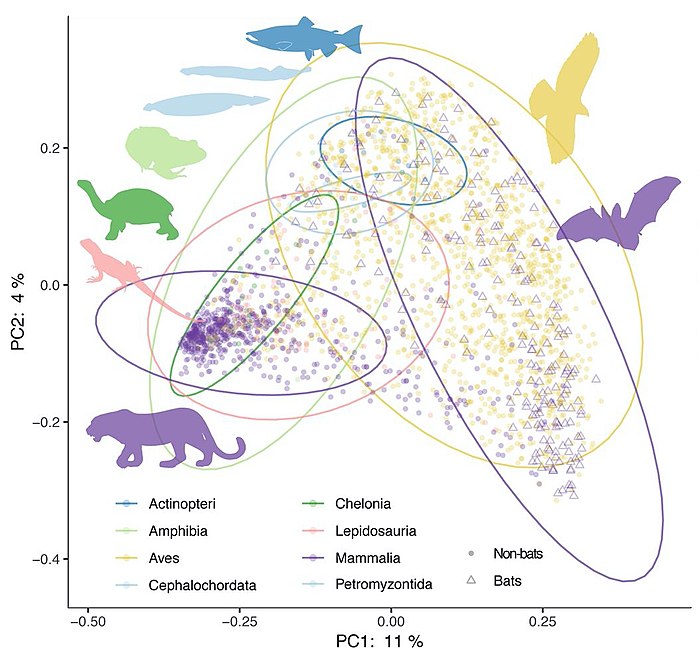
- PCoA analýza vytvořená z dat střevního mikrobiomu zvířat. Šablona:Hsp[113]
Střevní mikrobiom savců je klíčovým regulátorem fyziologie hostitele.[114] Koevoluce mezi hostitelem a mikroorganismy hrála klíčovou roli v adaptaci savců na jejich různorodý životní styl. Strava, zejména pak býložravost, je důležitým korelačním prvkem mikrobiální diverzity u savců.[115][116] Většina savčích mikrobiomů také silně koreluje s fylogenezí hostitele.[115][117][118][119] To naznačuje, že hostitelské faktory, které se mění napříč hostitelskou fylogenezí (např. fyziologie střev) hrají důležitou roli při strukturování střevních mikrobiomů u savců. O adaptivním imunitním systému obratlovců se dokonce spekuluje, že se vyvinul jako právě takový faktor pro selektivní udržování symbiotické homeostázy.[120]
Významnost jednotlivých faktorů souvisejících s fylogenezí pro diverzitu mikrobiomů obratlovců je stále velmi málo objasněn. Fylosymbióza neboli stav, kdy blíže příbuzné hostitelské druhy mají více podobný mikrobiom,[121][122] byla popsána u řady druhů mimo savce.[123][124] Jiné studie přichází s protichůdnými výsledky a ukazují naopak na podstatné rozdíly ve fylosymbióze mezi taxony savců.[125][126][127] Pokud je zaznamenána přítomnost robustní fylosymbiotické korelace, znamená to, že hostitelské faktory řídí a kontrolují vytváření svých mikrobiálních komunit a jejich ustalování. I když jsou specifické mechanismy kontroly hostitelským organismem neznámé, variace v intenzitě nebo přítomnosti měřitelného fylosymbiotického signálu napříč hostitelskou fylogenezí se jeví jako užitečná pro identifikaci a objasnění takových mechanismů.
Člověk
Lidský mikrobiom je souhrn veškeré mikrobioty, která sídlí vně nebo uvnitř lidských tkáních a biologických tekutinách[128] včetně kůže, mléčných žláz, semenné tekutiny, dělohy, ovariálních folikulů, plic, slin a sliznice dutiny ústní, spojivky, žlučových cest a gastrointestinálního traktu. Mezi typy lidské mikrobioty patří bakterie, archea, houby, protozoa a viry. Přestože mikroskopičtí živočichové mohou také žít na lidském těle, jsou z této definice obecně vyloučeni. V kontextu genomiky se termín lidský mikrobiom někdy používá k označení kolektivních genomů rezidentních mikroorganismů;[129] stejný význam má i termín lidský metagenom.[128]
Lidé tělo je kolonizováno mnoha mikroorganismy, přičemž hodnoty lidských a nelidských buněk jsou řádově přibližně stejné.[130] Některé mikroorganismy kolonizující člověka jsou komenzální, což znamená, že s člověkem jakožto hostujícím organismem koexistují, aniž by mu škodily nebo prospívaly. Jiní mají se svými lidskými hostiteli mutualistický vztah.[129] :s.700[131] Některé nepatogenní mikroorganismy mohou naopak poškodit lidské hostitele prostřednictvím metabolitů, které produkují. Příkladem může být trimethylamin, který lidské tělo přeměňuje na trimethylamin N-oxid oxidací zprostředkovanou enzymem FMO3.[132][133] Některé mikroorganismy vykonávají úlohy užitečné pro lidského hostitele. Ty, u kterých se očekává, že budou přítomny, a které za normálních okolností nezpůsobují onemocnění, jsou považovány za normální mikrobiotu.[129] Složení zdravého lidského mikrobiomu přesto není dosud definováno.
Masivní projekt zaměřený na lidský mikrobiom (HMP-Human Microbiome Project) si pojal za cíl sekvenování genomu lidské mikrobioty se zaměřením zejména na mikrobiotu, která normálně obývá kůži, ústa, nos, trávicí trakt a vagínu.[129] Milníku dosáhla v roce 2012, kdy zveřejnila své první výsledky.[134]
V roce 2016 Bashan s kolektivem zjistili, že ačkoli různí lidé mají různé mikrobiální druhy s různou abundancí, dynamika lidských mikrobiálních společenství není unikátní, ale naopak se mikrobiota každého řídí velmi podobnými pravidly. Zjištění má význam pro pochopení zdraví a nemoci a pomáhá pochopit, jak se mikrobiální komunity chovají a mění v čase.[135]
Dostupné metody pro studium mikrobiomů, tzv. multiomiky, pokrývají celou oblast studia od izolace, kultivace (kulturomika) a vizualizace (mikroskopie), až po zacílení na taxonomické složení (metabarcoding) nebo řešení metabolického potenciálu (tzv. metabarkódování funkčních genů, metagenomika) a také dokáží analyzovat mikrobiální aktivitu (metatranskriptomika, metaproteomika, metabolomika). Na základě metagenomových dat lze rekonstruovat přítomné mikrobiální genomy. První genomy sestavené z metagenomu byly rekonstruovány ze vzorků životního prostředí[136] a v posledních letech bylo touto metodou získáno již několik tisíc bakteriálních genomů bez nutnosti kultivace vzorku. Například 154 723 mikrobiálních genomů globálního lidského mikrobiomu bylo v roce 2019 rekonstruováno z celkem 9 428 metagenomů.[137]
Počítačová simulace je nástroj, který se při studiu mikrobiomů využívá jako doplnění k experimentálním metodám a slouží pro zkoumání mikrobiálních funkcí využitím multi-omických dat a k predikci komplexní mezidruhové a hostitelsko-druhové dynamiky.[138][139] Populární metodou in silico je kombinovat modely metabolické sítě mikrobiálních taxonů přítomných v komunitě a používat strategii matematického modelování, jako je metabolická flux analýza (tzv. flux balance analýza), k předpovědi metabolické funkce mikrobiální komunity na úrovni taxonu.[37][140]
Od roku 2020 nedochází k výraznému posunu v objasnění problematiky, a to především kvůli konfliktu mezi masivní dostupností dat o sekvencích DNA mikrobiomu na jedné straně a omezenou dostupností mikrobiálních izolátů potřebných k potvrzení metagenomických předpovědí funkce genu na straně druhé. I když metagenomová data poskytují prostor pro nové predikce a hypotézy, je k posílení vazeb mezi sekvencí a přísnými funkčními předpověďmi zapotřebí mnohem více dat. Toto je zřejmé v situaci, když se vezme v úvahu, že nahrazení jedné jediné aminokyseliny jinou může vést k radikální funkční změně, která tudíž vede k nesprávnému přiřazení funkce k dané genové sekvenci.[141] Kromě toho je zapotřebí pracovat na technikách umožňujících kultivaci nových kmenů, která pomůže identifikovat velkou část neznámých sekvencí získaných z metagenomických analýz. Takových může být u nedostatečně prozkoumaných ekosystémů více než 70 %. V závislosti na použité metodě sekvenace, dokonce i v dobře prostudovaných mikrobiomech, 40–70 % anotovaných genů v plně sekvenovaných mikrobiálních genomech nemá žádnou známou nebo předpokládanou funkci.[142] Do roku 2019 nemělo 85 z tehdy zavedených 118 kmenů popsán jediný bakteriální druh, což představuje výzvu k pochopení prokaryotické funkční diverzity.[143][1]
Počet prokaryotických kmenů může dosáhnout stovek, zatímco doména archea je nejméně prozkoumaná.[143] Zvyšující se rozdíl mezi rozmanitostí kultivovatelných bakterií a archeí a těmi, které byly zjištěny molekulárními metodami, vedla k návrhu vytvořit formální nomenklaturu pro dosud nekultivované taxony, primárně založenou na sekvenčních informacích.[144][145]
- Základní popis problematiky
- Kompromisy mezi experimentálními otázkami a složitostí systémů mikrobiomů[146]
(A) Vzájemné interakce mezi půdními bakteriemi Bacillus subtilis a Streptomyces spp. se hodí pro charakterizaci funkcí sekundárních metabolitů v mikrobiálních interakcích.
(B) Symbióza mezi olihní a mořskou bakterií Aliivibrio fischeri je zásadní pro pochopení hostitelských a mikrobiálních faktorů, které ovlivňují kolonizaci.
(C) Použití modelu gnotobiotických myší pro mikrobiální studie je zásadní pro vytvoření spojení mezi stravou hostitele a účinky na specifické mikrobiální taxony v komunitě.[146]
Reference
V tomto článku byl použit překlad textu z článku Microbiome na anglické Wikipedii.
- ↑ a b c d e f g h i Chybí název periodika! PMID 32605663. Bibcode 4..
- ↑ Merchak A, Gaultier A. Microbial metabolites and immune regulation: New targets for major depressive disorder. Brain Behav Immun Health. 2020 Nov 2;9:100169. doi: 10.1016/j.bbih.2020.100169. PMID 34589904; PMCID: PMC8474524.
- ↑ Hiltner L. (1902) "Die Keimungsverhältnisse der Leguminosensamen und ihre Beeinflussung durch Organismenwirkung". In: Parey P and Springer J (Eds.) Arb Biol Abt Land u Forstw K Gsndhtsamt, 3, Berlin. Pages 1–545.
- ↑ Metchnikoff E. The prolongation of life: optimistic studies. GP Putnam's Sons; 1908.
- ↑ Bassler, B.L. (2002) "Small talk: cell-to-cell communication in bacteria". Cell, 109(4): 421–424. DOI:10.1016/S0092-8674(02)00749-3.
- ↑ Brul, S., Kallemeijn, W. and Smits, G. (2008) "Functional genomics for food microbiology: molecular mechanisms of weak organic-acid preservative adaptation in yeast". CAB Rev, 3: 1–14. DOI:10.1079/PAVSNNR20083005.
- ↑ a b Chybí název periodika! PMID 270744.
- ↑ Uksa, M., Schloter, M., Endesfelder, D., Kublik, S., Engel, M., Kautz, T., Köpke, U. and Fischer, D. (2015) "Prokaryotes in subsoil—evidence for a strong spatial separation of different phyla by analysing co-occurrence networks". Frontiers in microbiology, 6: 1269. DOI:10.3389/fmicb.2015.01269.
- ↑ Maritz, J.M., Rogers, K.H., Rock, T.M., Liu, N., Joseph, S., Land, K.M. and Carlton, J.M. (2017) "An 18S rRNA workflow for characterizing protists in sewage, with a focus on zoonotic trichomonads". Microbial ecology, 74(4): 923–936. DOI:10.1007/s00248-017-0996-9.
- ↑ Purahong, W., Wubet, T., Lentendu, G., Schloter, M., Pecyna, M.J., Kapturska, D., Hofrichter, M., Krüger, D. and Buscot, F. (2016) "Life in leaf litter: novel insights into community dynamics of bacteria and fungi during litter decomposition". Molecular Ecology, 25(16): 4059–4074. DOI:10.1111/mec.13739.
- ↑ Lozupone, C.A., Stombaugh, J.I., Gordon, J.I., Jansson, J.K. and Knight, R. (2012) "Diversity, stability and resilience of the human gut microbiota". Nature, 489(7415): 220–230. DOI:10.1038/nature11550.
- ↑ Venter, J.C., Remington, K., Heidelberg, J.F., Halpern, A.L., Rusch, D., Eisen, J.A., Wu, D., Paulsen, I., Nelson, K.E., Nelson, W. and Fouts, D.E. (2004) "Environmental genome shotgun sequencing of the Sargasso Sea". Science, 304(5667): 66–74. DOI:10.1126/science.1093857.
- ↑ Liu, L., Li, Y., Li, S., Hu, N., He, Y., Pong, R., Lin, D., Lu, L. and Law, M. (2012) "Comparison of next-generation sequencing systems". BioMed Research International, 2012: 251364. DOI:10.1155/2012/251364.
- ↑ Stegen, J.C., Bottos, E.M. and Jansson, J.K. (2018) "A unified conceptual framework for prediction and control of microbiomes". Current Opinion in Microbiology, 44: 20–27. DOI:10.1016/j.mib.2018.06.002.
- ↑ Knight, R., Vrbanac, A., Taylor, B.C., Aksenov, A., Callewaert, C., Debelius, J., Gonzalez, A., Kosciolek, T., McCall, L.I., McDonald, D. and Melnik, A.V. (2018) "Best practices for analysing microbiomes". Nature Reviews Microbiology, 16(7): 410–422. DOI:10.1038/s41579-018-0029-9.
- ↑ Chybí název periodika! PMID 25750239.
- ↑ Chybí název periodika! PMID 16200144.
- ↑ Chybí název periodika! PMID 22882766.
- ↑ conservancy.umn.edu. Dostupné online. PMID 22092289.
- ↑ Dostupné online.
- ↑ Chybí název periodika! PMID 13076173.
- ↑ Chybí název periodika! PMID 20474956.
- ↑ Chybí název periodika! PMID 33226.
- ↑ Chybí název periodika! PMID 28980196.
- ↑ Chybí název periodika! PMID 12660721.
- ↑ Chybí název periodika! PMID 24747923.
- ↑ Chybí název periodika! PMID 4913914.
- ↑ [s.l.]: [s.n.] Dostupné online. ISBN 978-1-118-68134-3. OCLC 864917338
- ↑ Chybí název periodika! PMID 1105573.
- ↑ Chybí název periodika! PMID 271968.
- ↑ discovery.ucl.ac.uk. Dostupné online. PMID 26554401.
- ↑ Chybí název periodika! PMID 29123225.
- ↑ Chybí název periodika! PMID 1264203.
- ↑ Chybí název periodika! PMID 6801762.
- ↑ Chybí název periodika! PMID 7764001.
- ↑ Chybí název periodika! PMID 2830981.
- ↑ a b c Chybí název periodika!
- ↑ [s.l.]: [s.n.] Dostupné online. ISBN 978-0-262-13269-5. OCLC 22597587
- ↑ Chybí název periodika! PMID 16470363.
- ↑ ; Flemming. [s.l.]: [s.n.] Dostupné online. ISBN 978-3-642-19940-0. OCLC 769756150
- ↑ Chybí název periodika! PMID 7535888.
- ↑ Chybí název periodika! PMID 7542800. Bibcode 1.
- ↑ [s.l.]: [s.n.] Dostupné online. ISBN 978-953-51-2240-1. DOI 10.5772/61964.
- ↑ Chybí název periodika! PMID 22732228.
- ↑ Chybí název periodika! PMID 20858600.
- ↑ Chybí název periodika! PMID 21304728.
- ↑ Konopka, A. (2009) "What is microbial community ecology?" The ISME Journal, 3(11): 1223–1230. Konopka, A., 2009. What is microbial community ecology?. The ISME journal, 3(11), pp.1223–1230. DOI:10.1038/ismej.2009.88.
- ↑ a b c d Whipps J., Lewis K. and Cooke R. (1988) "Mycoparasitism and plant disease control". In: Burge M (Ed.) Fungi in Biological Control Systems, Manchester University Press, pages 161–187. ISBN 9780719019791.
- ↑ a b Lederberg, J. and McCray, A.T. (2001) "'Ome Sweet'Omics—A genealogical treasury of words". The Scientist, 15(7): 8.
- ↑ a b c d e Marchesi, J.R. and Ravel, J. (2015) "The vocabulary of microbiome research: a proposal". Microbiome, 3(31). DOI:10.1186/s40168-015-0094-5.
- ↑ Prosser, J.I., Bohannan, B.J., Curtis, T.P., Ellis, R.J., Firestone, M.K., Freckleton, R.P., Green, J.L., Green, L.E., Killham, K., Lennon, J.J. and Osborn, A.M. (2007) "The role of ecological theory in microbial ecology". Nature Reviews Microbiology, 5(5): 384–392. DOI:10.1038/nrmicro1643.
- ↑ del Carmen Orozco-Mosqueda, M., del Carmen Rocha-Granados, M., Glick, B.R. and Santoyo, G. (2018) "Microbiome engineering to improve biocontrol and plant growth-promoting mechanisms". Microbiological Research, 208: 25–31. DOI:10.1016/j.micres.2018.01.005.
- ↑ a b Merriam-Webster Dictionary – microbiome.
- ↑ Human Microbiome Project. Accessed 25 Aug 2020.
- ↑ Nature.com: Microbiome. Accessed 25 August 2020.
- ↑ ScienceDirect: Microbiome Accessed 25 August 2020.
- ↑ Arevalo, P., VanInsberghe, D., Elsherbini, J., Gore, J. and Polz, M.F. (2019) "A reverse ecology approach based on a biological definition of microbial populations". Cell, 178(4): 820–834. DOI:10.1016/j.cell.2019.06.033.
- ↑ Schlaeppi, K. and Bulgarelli, D. (2015) "The plant microbiome at work". Molecular Plant-Microbe Interactions, 28(3): 212–217. DOI:10.1094/MPMI-10-14-0334-FI.
- ↑ Rogers Y-H and Zhang C. (2016) "Genomic Technologies in Medicine and Health: Past, Present, and Future". In: Kumar D and Antonarakis S. (Eds.) Medical and Health Genomics. Oxford: Academic Press, pages 15–28. ISBN 9780127999227.
- ↑ Ho, H.E. and Bunyavanich, S. (2018) "Role of the microbiome in food allergy". Current allergy and asthma reports, 18(4): 27. DOI:10.1007/s11882-018-0780-z.
- ↑ Whiteside, S.A., Razvi, H., Dave, S., Reid, G. and Burton and J.P. (2015) "The microbiome of the urinary tract—a role beyond infection". Nature Reviews Urology, 12(2): 81–90. DOI:10.1038/nrurol.2014.361.
- ↑ MicrobiomeSupport project
- ↑ Carini, Paul (2016) A census of the dead: the story behind microbial ‘relic DNA’ in soil Archivováno 28. 9. 2021 na Wayback Machine. Nature Research: Microbiology.
- ↑ Carini, P., Marsden, P.J., Leff, J.W., Morgan, E.E., Strickland, M.S. and Fierer, N. (2016) "Relic DNA is abundant in soil and obscures estimates of soil microbial diversity". Nature Microbiology, 2(3): 1–6. DOI:10.1038/nmicrobiol.2016.242.
- ↑ a b Lennon, J.T., Muscarella, M.E., Placella, S.A. and Lehmkuhl, B.K. (2018) "How, when, and where relic DNA affects microbial diversity". mBio, 9(3). DOI:10.1128/mBio.00637-18.
- ↑ Dupré JO, O’Malley MA (2009) "Varieties of living things: life at the intersection of lineage and metabolism". In: Normandin S and Wolfe C (Eds.) Vitalism and the Scientific Image in Post-Enlightenment Life Science 1800–2010. Dordrecht: Springer, pages 311–344. ISBN 9789400724457.
- ↑ McDaniel, L., Breitbart, M., Mobberley, J., Long, A., Haynes, M., Rohwer, F. and Paul, J.H., 2008. Metagenomic analysis of lysogeny in Tampa Bay: implications for prophage gene expression. PLoS One, 3(9), p.e3263. DOI:10.1371/journal.pone.0003263.
- ↑ www.zora.uzh.ch. Dostupné online. PMID 29789680.
- ↑ RIERA, Joan Lluís; BALDO, Laura. Microbial co-occurrence networks of gut microbiota reveal community conservation and diet-associated shifts in cichlid fishes. Animal Microbiome. Springer Science and Business Media LLC, 29 September 2020, s. 36. ISSN 2524-4671. DOI 10.1186/s42523-020-00054-4. PMID 33499972.
- ↑ Chybí název periodika! PMID 11462219.
- ↑ Chybí název periodika! PMID 28115400.
- ↑ Chybí název periodika! PMID 32873208.
- ↑ Apprill, A. (2017) "Marine animal microbiomes: toward understanding host–microbiome interactions in a changing ocean". Frontiers in Marine Science, 4: 222. DOI:10.3389/fmars.2017.00222.
- ↑ Webster, N.S., Negri, A.P., Botté, E.S., Laffy, P.W., Flores, F., Noonan, S., Schmidt, C. and Uthicke, S. (2016) "Host-associated coral reef microbes respond to the cumulative pressures of ocean warming and ocean acidification". Scientific reports, 6: 19324. DOI:10.1038/srep19324.
- ↑ Daniels, C. and Breitbart, M. (2012) "Bacterial communities associated with the ctenophores Mnemiopsis leidyi and Beroe ovata". FEMS Microbiology Ecology, 82(1): 90–101. DOI:10.1111/j.1574-6941.2012.01409.x.
- ↑ Blasiak, L.C., Zinder, S.H., Buckley, D.H. and Hill, R.T. (2014) "Bacterial diversity associated with the tunic of the model chordate Ciona intestinalis". The ISME Journal, 8(2): 309–320. DOI:10.1038/ismej.2013.156.
- ↑ Givens, C.E., Ransom, B., Bano, N. and Hollibaugh, J.T. (2015) "Comparison of the gut microbiomes of 12 bony fish and 3 shark species". Marine Ecology Progress Series, 518: 209–223. DOI:10.3354/meps11034.
- ↑ McFall-Ngai, M.J. (2000) "Negotiations between animals and bacteria: the ‘diplomacy’of the squid-vibrio symbiosis". Comparative Biochemistry and Physiology, Part A: Molecular & Integrative Physiology, 126(4): 471–480. DOI:10.1016/S1095-6433(00)00233-6.
- ↑ McFall-Ngai, M. (2014) "Divining the essence of symbiosis: insights from the squid-vibrio model". PLoS Biology, 12(2): e1001783. DOI:10.1371/journal.pbio.1001783.
- ↑ Dubilier, N., Mülders, C., Ferdelman, T., de Beer, D., Pernthaler, A., Klein, M., Wagner, M., Erséus, C., Thiermann, F., Krieger, J. and Giere, O. (2001) "Endosymbiotic sulphate-reducing and sulphide-oxidizing bacteria in an oligochaete worm". Nature, 411(6835): 298–302. DOI:10.1038/35077067.
- ↑ Schimak, M.P., Kleiner, M., Wetzel, S., Liebeke, M., Dubilier, N. and Fuchs, B.M. (2016) "MiL-FISH: Multilabeled oligonucleotides for fluorescence in situ hybridization improve visualization of bacterial cells". Applied and Environmental Microbiology, 82(1): 62–70. DOI:10.1128/AEM.02776-15.
- ↑ Kleiner, M., Wentrup, C., Lott, C., Teeling, H., Wetzel, S., Young, J., Chang, Y.J., Shah, M., VerBerkmoes, N.C., Zarzycki, J. and Fuchs, G. (2012) "Metaproteomics of a gutless marine worm and its symbiotic microbial community reveal unusual pathways for carbon and energy use". Proceedings of the National Academy of Sciences, 109(19): E1173–E1182. DOI:10.1073/pnas.1121198109.
- ↑ Wippler, J., Kleiner, M., Lott, C., Gruhl, A., Abraham, P.E., Giannone, R.J., Young, J.C., Hettich, R.L. and Dubilier, N. (2016) "Transcriptomic and proteomic insights into innate immunity and adaptations to a symbiotic lifestyle in the gutless marine worm Olavius algarvensis". BMC Genomics, 17(1): 942. DOI:10.1186/s12864-016-3293-y.
- ↑ Woyke, T., Teeling, H., Ivanova, N.N., Huntemann, M., Richter, M., Gloeckner, F.O., Boffelli, D., Anderson, I.J., Barry, K.W., Shapiro, H.J. and Szeto, E. (2006) "Symbiosis insights through metagenomic analysis of a microbial consortium". Nature, 443(7114): 950–955. DOI:10.1038/nature05192.
- ↑ Ruehland, C., Blazejak, A., Lott, C., Loy, A., Erséus, C. and Dubilier, N. (2008) "Multiple bacterial symbionts in two species of co‐occurring gutless oligochaete worms from Mediterranean sea grass sediments". Environmental microbiology, 10(12): 3404–3416. DOI:10.1111/j.1462-2920.2008.01728.x.
- ↑ a b Neave, M.J., Apprill, A., Ferrier-Pagès, C. and Voolstra, C.R. (2016) "Diversity and function of prevalent symbiotic marine bacteria in the genus Endozoicomonas". Applied Microbiology and Biotechnology, 100(19): 8315–8324. DOI:10.1007/s00253-016-7777-0.
- ↑ Dubinsky, Z. and Jokiel, P.L. (1994) "Ratio of energy and nutrient fluxes regulates symbiosis between zooxanthellae and corals". Pacific Science, 48(3): 313–324.
- ↑ Anthony, K.R., Kline, D.I., Diaz-Pulido, G., Dove, S. and Hoegh-Guldberg, O.(2008) "Ocean acidification causes bleaching and productivity loss in coral reef builders". Proceedings of the National Academy of Sciences, 105(45): 17442–17446. DOI:10.1073/pnas.0804478105.
- ↑ Bourne, D.G., Morrow, K.M. and Webster, N.S. (2016) "Insights into the coral microbiome: underpinning the health and resilience of reef ecosystems". Annual Review of Microbiology, 70: 317–340. DOI:10.1146/annurev-micro-102215-095440.
- ↑ Neave, M.J., Michell, C.T., Apprill, A. and Voolstra, C.R. (2017) "Endozoicomonas genomes reveal functional adaptation and plasticity in bacterial strains symbiotically associated with diverse marine hosts". Scientific Reports, 7: 40579. DOI:10.1038/srep40579.
- ↑ Hughes, T.P., Kerry, J.T., Álvarez-Noriega, M., Álvarez-Romero, J.G., Anderson, K.D., Baird, A.H., Babcock, R.C., Beger, M., Bellwood, D.R., Berkelmans, R. and Bridge, T.C. (2017) "Global warming and recurrent mass bleaching of corals". Nature, 543(7645): 373–377. DOI:10.1038/nature21707.
- ↑ Bell, J.J. (2008) "The functional roles of marine sponges". Estuarine, Coastal and Shelf Science, 79(3): 341–353. DOI:10.1016/j.ecss.2008.05.002.
- ↑ Webster, N.S. and Thomas, T. (2016) "The sponge hologenome". mBio, 7(2). DOI:10.1128/mBio.00135-16.
- ↑ Bayer, K., Schmitt, S. and Hentschel, U. (2008) "Physiology, phylogeny and in situ evidence for bacterial and archaeal nitrifiers in the marine sponge Aplysina aerophoba". Environmental Microbiology, 10(11): 2942–2955. DOI:10.1111/j.1462-2920.2008.01582.x.
- ↑ Radax, R., Hoffmann, F., Rapp, H.T., Leininger, S. and Schleper, C. (2012) "Ammonia‐oxidizing archaea as main drivers of nitrification in cold‐water sponges". Environmental Microbiology, 14(4): 909_923. DOI:10.1111/j.1462-2920.2011.02661.x.
- ↑ Zhang, F., Blasiak, L.C., Karolin, J.O., Powell, R.J., Geddes, C.D. and Hill, R.T. (2015) "Phosphorus sequestration in the form of polyphosphate by microbial symbionts in marine sponges". Proceedings of the National Academy of Sciences, 112(14): 4381–4386. DOI:10.1073/pnas.1423768112.
- ↑ Colman, A.S. (2015) "Sponge symbionts and the marine P cycle". Proceedings of the National Academy of Sciences, 112(14): 4191–4192. DOI:10.1073/pnas.1502763112.
- ↑ Simister, R., Taylor, M.W., Tsai, P., Fan, L., Bruxner, T.J., Crowe, M.L. and Webster, N. (2012) "Thermal stress responses in the bacterial biosphere of the Great Barrier Reef sponge, Rhopaloeides odorabile. Environmental Microbiology, 14(12): 3232–3246. DOI:10.1111/1462-2920.12010.
- ↑ Morrow, K.M., Bourne, D.G., Humphrey, C., Botté, E.S., Laffy, P., Zaneveld, J., Uthicke, S., Fabricius, K.E. and Webster, N.S. (2015) "Natural volcanic CO 2 seeps reveal future trajectories for host–microbial associations in corals and sponges". The ISME Journal, 9(4): 894–908. DOI:10.1038/ismej.2014.188.
- ↑ Ribes, M., Calvo, E., Movilla, J., Logares, R., Coma, R. and Pelejero, C. (2016) "Restructuring of the sponge microbiome favors tolerance to ocean acidification. Environmental Microbiology Reports, 8(4): 536–544. DOI:10.1111/1758-2229.12430.
- ↑ Acevedo-Whitehouse K, Rocha-Gosselin A, Gendron D. A novel non‐invasive tool for disease surveillance of free‐ranging whales and its relevance to conservation programs.. Animal Conservation. April 2010, s. 217–225. DOI 10.1111/j.1469-1795.2009.00326.x. S2CID 86518859.
- ↑ Pirotta V, Smith A, Ostrowski M, Russell D, Jonsen ID, Grech A, Harcourt R. An economical custom-built drone for assessing whale health.. Frontiers in Marine Science. December 2017, s. 425. DOI 10.3389/fmars.2017.00425.
- ↑ Chybí název periodika! PMID 31210263.
- ↑ Chybí název periodika! PMID 23761254.
- ↑ Chybí název periodika! PMID 31260051.
- ↑ Chybí název periodika! PMID 32614926.
- ↑ Chybí název periodika! PMID 29865228.
- ↑ a b Chybí název periodika! PMID 23805896.
- ↑ Compant, S., Samad, A., Faist, H. and Sessitsch, A. (2019) "A review on the plant microbiome: Ecology, functions, and emerging trends in microbial application". Journal of advanced research, 19: 29_37.DOI:10.1016/j.jare.2019.03.004.
- ↑ Chybí název periodika! PMID 26052316.
- ↑ Chybí název periodika! PMID 22564542.
- ↑ Chybí název periodika! PMID 25278934.
- ↑ SONG, Se Jin; SANDERS, Jon G.; DELSUC, Frédéric; METCALF, Jessica; AMATO, Katherine; TAYLOR, Michael W.; MAZEL, Florent. Comparative Analyses of Vertebrate Gut Microbiomes Reveal Convergence between Birds and Bats. mBio. 2020. DOI 10.1128/mBio.02901-19. PMID 31911491.
- ↑ Chybí název periodika! PMID 23391737. Bibcode 4.
- ↑ a b Chybí název periodika! PMID 18497261.
- ↑ Chybí název periodika! PMID 21596990.
- ↑ Chybí název periodika! PMID 29995839.
- ↑ Chybí název periodika! PMID 28230052.
- ↑ Chybí název periodika! PMID 26393325.
- ↑ Chybí název periodika! PMID 17215830.
- ↑ Chybí název periodika! PMID 22276533.
- ↑ Chybí název periodika! PMID 27861590.
- ↑ Chybí název periodika! PMID 24304129.
- ↑ Chybí název periodika! PMID 30467310.
- ↑ Chybí název periodika! PMID 29290090.
- ↑ Chybí název periodika! PMID 22519571. Bibcode 4.
- ↑ Chybí název periodika! PMID 26042099.
- ↑ a b Chybí název periodika! PMID 26229597.
- ↑ a b c d [s.l.]: [s.n.] Dostupné online. ISBN 9780073402406. OCLC 886600661
- ↑ Chybí název periodika! PMID 26824647.
- ↑ Chybí název periodika! PMID 24729765.
- ↑ Chybí název periodika! PMID 26274026.
- ↑ Chybí název periodika! PMID 25473158.
- ↑ Dostupné online.
- ↑ www.nature.com. Dostupné online. PMID 27279224.
- ↑ Chybí název periodika! PMID 27774985. Bibcode 4.
- ↑ Chybí název periodika! PMID 30661755. Bibcode 4.
- ↑ dx.doi.org. Dostupné online. PMID 31337891.
- ↑ doi.org. Dostupné online. PMID 22589385.
- ↑ dx.doi.org. Dostupné online. PMID 26109480.
- ↑ Chybí název periodika! PMID 19528653.
- ↑ Chybí název periodika! PMID 29173869.
- ↑ a b Chybí název periodika! PMID 30197212.
- ↑ Chybí název periodika! PMID 28731467.
- ↑ Chybí název periodika! PMID 30098831.
- ↑ a b Chevrette, M.G., Bratburd, J.R., Currie, C.R. and Stubbendieck, R.M. (2019 "Experimental Microbiomes: Models Not to Scale". mSystems, 4(4): e00175-19. DOI:10.1128/mSystems.00175-19.
Související články
Média použitá na této stránce
Autor:
- Emblem-web.svg: David Vignoni
- Broom icon.svg: User:Booyabazooka (uploader)
- derivative work: Ju gatsu mikka (^o^) appelez moi Ju (^o^)
Web broom icon
Autor: Gabriele Berg, Daria Rybakova and 33 others, Licence: CC BY-SA 4.0
Composition of the term microbiome containing both the microbiota (community of microorganisms) and their “theatre of activity” (structural elements, metabolites/signal molecules, and the surrounding environmental conditions)
Autor: Gabriele Berg, Daria Rybakova and 33 others, Licence: CC BY-SA 4.0
Microbial interactions visualized through microbial co-occurrence networks.
a) Microbial interactions are influenced by environmental factors and are separated into positive, neutral, and negative interactions types. b) Microbial co-occurrence and co-exclusion networks help visualizing microbial interactions. In such networks, nodes usually represent taxa of microorganisms, and edges represent statistically significant associations between nodes. Green edges usually stay for positive interactions, while red edges visualize negative interactions between the microorganisms. c) Testing of the hypotheses resulted from the network analyses in relevant model systems is required for a comprehensive study of microbial interactions
Autor: Song SJ, Sanders JG, Delsuc F, Metcalf J, Amato K, Taylor MW, Mazel F, Lutz HL, Winker K, Graves GR, Humphrey G., Licence: CC BY-SA 4.0
Principal-coordinate analysis of animal microbiome data sets
Principal-coordinate analysis of unweighted UniFrac distances between samples rarefied to 10,000 sequences/sample and filtered to include only up to 5 individuals per species (2,258 points). Colors represent host class, with the mammalian order Chiroptera shown as triangles; 95% confidence intervals per class represented by colored ellipses (separately for bats and nonflying mammals).
Autor: Amy Apprill, Licence: CC BY-SA 4.0
Conceptual diagram of a marine animal host-microbiome relationship. Relationships are generally thought to exist in a symbiotic state, and are normally exposed to environmental and animal-specific factors that may cause natural variations. Some events may change the relationship into a functioning but altered symbiotic state, whereas extreme stress events may cause dysbiosis or a breakdown of the relationship and interactions.
Autor: Pirotta V, Smith A, Ostrowski M, Russell D, Jonsen ID, Grech A and Harcourt R, Licence: CC BY-SA 4.0
cetacean blow's bacteria
Autor: Acevedo-Whitehouse, K, A Rocha-Gosselin, and D Gendron, Licence: CC BY-SA 4.0
whale blow sampling with drone
Autor: Joan Lluís Riera & Laura Baldo, Licence: CC BY-SA 4.0
Co-occurrence networks showing difference in gut microbiota between herbivorous and carnivorous cichlids
Diet-specific networks of Lake Tanganyika cichlid herbivores and carnivores. Nodes are colored according to phylum and sized by betweenness values normalized by lake. Grey shades represent distinct modules (connected by red edges). Notice the higher complexity of the herbivore network (156 nodes and 339 edges; 19 modules) compared to the carnivore network (21 nodes and 70 edges; 3 modules)
Autor: Matthew J. Neave, Amy Apprill, Christine Ferrier-Pagès and Christian R. Voolstra, Licence: CC BY-SA 4.0
Photograph of Stylophora pistillata colony from Nukuoro atoll, Federated States of Micronesia (a), and microscopic photo of Endozoicomonas (Ez) probed cells (yellow) within the tentacles of S. pistillata residing in aggregates (Ez agg) as well as just outside the aggregate (b). Endozoicomonas cells are hybridized with the horseradish peroxidase-labelled probe targeting the Endozoicomonas bacteria, and the blue staining is a general nucleic acid stain (DAPI)
Autor: Marc G. Chevrette, Jennifer R. Bratburd, Cameron R. Currie and Reed M. Stubbendieck. Specific original image credit from the Noun Project: Fertile Soil by Ben Davis; Droplet by Focus; Mouse by Iconic; Cheese Wheel by Anniken & Andreas; Bacteria by Arthur Shlain; Squid by Artem Kovyazin; ant by Yugudesign; leaf by Saeful Muslim, Licence: CC BY 3.0
Tradeoffs between experimental questions and complexity of microbiome systems.
Each microbiome system is suited to address different types of questions based on the culturability of microbes, genetic tractability of microbes and host (where relevant), ability to maintain system in laboratory setting, and ability to make host/environment germfree. Three different systems are shown in this figure as examples. (A) Pairwise interactions between B. subtilis and Streptomyces spp. are well-suited for characterizing the functions of secondary metabolites in microbial interactions. (B) The symbiosis between bobtail squid and A. fischeri is fundamental to understanding host and microbial factors that influence colonization. (C) The use of gnotobiotic mice is crucial for making links between host diet and the effects on specific microbial taxa in a community
Autor: Gabriele Berg, Daria Rybakova and 33 others, Licence: CC BY-SA 4.0
Highlighting the shift of the paradigm from microbes as unsocial organisms causing diseases to the holistic view of microorganisms being the center of the One Health Concept: positively interconnecting all areas of human lives.
Autor: Rahul Mahadev Shelake, Dibyajyoti Pramanik and Jae-Yean Kim, Licence: CC BY-SA 4.0
Microbiome in plant ecosystem
Schematic plant and plant-associated microbiota colonizing different niches on and inside the plant tissue. All the above-ground plant parts together, called the phyllosphere, are a continuously evolving habitat due to ultraviolet (UV) radiation and altering climatic conditions. It is primarily composed of leaves. Below-ground plant parts, mainly roots, are generally influenced by soil properties. Harmful interactions affect the plant growth through pathogenic activities of some microbiota members (left side). On the other hand, beneficial microbial interactions promote plant growth (right side).
![Posun paradigmatu směrem od mikrobů jakožto samostatných organismů způsobujících nemoci k holistickému pohledu na mikroorganismy jako na komunitu propojující všechny oblasti lidského života.[1]](http://upload.wikimedia.org/wikipedia/commons/3/33/Microbiome_paradigm_shifts.png)
![Sítě společného výskytu pomáhají vizualizovat mikrobiální interakce Uzly (kolečka) obvykle představují taxony mikroorganismů a čáry představují statisticky významné asociace mezi uzly.[1] Pro komplexní studium mikrobiálních interakcí je nutné testování hypotéz vyplývajících ze síťových analýz.[1]](http://upload.wikimedia.org/wikipedia/commons/thumb/2/21/Microbial_interactions_visualized_through_microbial_co-occurrence_networks.webp/615px-Microbial_interactions_visualized_through_microbial_co-occurrence_networks.webp.png)
![Sítě společného výskytu ukazují rozdíl ve střevní mikrobiotě mezi býložravými a masožravými cichlidami Uzly (kolečka) jsou zbarvené podle kmene. Síť býložravců (Herbivores) má vyšší komplexitu (156 uzlů a 339 propojení) ve srovnání se sítí masožravců (Carnivores) (21 uzlů a 70 propojení).[69]](http://upload.wikimedia.org/wikipedia/commons/thumb/d/d0/Co-occurrence_networks_showing_difference_in_gut_microbiota_between_herbivorous_and_carnivorous_cichlids.webp/562px-Co-occurrence_networks_showing_difference_in_gut_microbiota_between_herbivorous_and_carnivorous_cichlids.webp.png)
![Obecně se předpokládá, že vztahy v rámci mořského mikrobiomu jsou v symbiotickém stavu a jsou normálně vystaveny environmentálním a zvířecím specifickým faktorům způsobující přirozené variace. Některé události mohou změnit vztah ve stále fungující, ale již změněný symbiotický stav, zatímco extrémní stresové události mohou způsobit dysbiózu nebo rozpad vztahu a interakcí.[73]](http://upload.wikimedia.org/wikipedia/commons/b/be/Marine_animal_host-microbiome_relationships.jpg)
![Odběr vzorku z proudu vody tryskajícího po vynoření modré velryby pomocí dronu vrtulníku[101]](http://upload.wikimedia.org/wikipedia/commons/thumb/f/f6/Whale_blow_sampling_with_drone.png/354px-Whale_blow_sampling_with_drone.png)
![Relativní množství bakteriálních tříd ze vzorků velryb, vzduchu a mořské vody.[102]](http://upload.wikimedia.org/wikipedia/commons/thumb/b/b5/Cetacean_blow%27s_bacteria.png/426px-Cetacean_blow%27s_bacteria.png)

![Kompromisy mezi experimentálními otázkami a složitostí systémů mikrobiomů[146] (A) Vzájemné interakce mezi půdními bakteriemi Bacillus subtilis a Streptomyces spp. se hodí pro charakterizaci funkcí sekundárních metabolitů v mikrobiálních interakcích. (B) Symbióza mezi olihní a mořskou bakterií Aliivibrio fischeri je zásadní pro pochopení hostitelských a mikrobiálních faktorů, které ovlivňují kolonizaci. (C) Použití modelu gnotobiotických myší pro mikrobiální studie je zásadní pro vytvoření spojení mezi stravou hostitele a účinky na specifické mikrobiální taxony v komunitě.[146]](http://upload.wikimedia.org/wikipedia/commons/thumb/0/03/Tradeoffs_between_experimental_questions_and_complexity_of_microbiome_systems.jpg/1054px-Tradeoffs_between_experimental_questions_and_complexity_of_microbiome_systems.jpg)











