Pyrosekvenování
Metoda pyrosekvenování spojuje metodu sekvenování využívající syntézu komplementárního vlákna pomocí DNA polymerázy (za použití primerů) a zároveň analýzu její aktivity pomocí dalších enzymů a chemikálií. Metoda je odvozená od Sangerovy metody sekvenování, využívá však reverzibilní terminaci polymerace. V roce 1987, P. Nyre´ popsal, jak aktivita DNA polymerázy může být monitorována pomocí bioluminiscence. [1]
Metoda
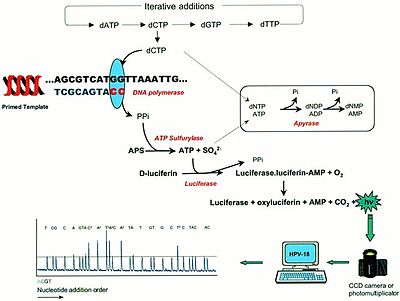
Systém pro pyrosekvenování je založený na práci 4 enzymů – DNA polymerázy I (nebo její Klenowův fragment), ATP sulfurylázy, luciferázy a apyrázy. Reakční směs dále obsahuje enzymatické substráty – adenosin fosfosulfát (APS), d-luciferin a sekvenovaný templát s příslušnými primery, na kterých může DNA polymeráza začít syntézu. Jednotlivé nukleotidy jsou přidávány postupně jeden po druhém.[1]
Postup
První reakcí je DNA polymerace pomocí DNA polymerázy, kdy dochází k zařazení příslušného deoxynukleotid trifosfátu (dNTPs) za uvolnění pyrofosfátu.
- (DNA)n + dNTP → (DNA)n+1 + PPi (polymeráza)
Vzniklý pyrofosfát je uvolněn z polymerázy a může sloužit jako substrát pro ATP sulfurylázu. Při této reakci dojde ke kvantitativnímu převedení pyrofosfátu na ATP.
- PPi + APS → ATP + SO42- (ATP sulfuryláza)
Během třetí a čtvrté reakce je ATP převedeno na světelný signál pomocí enzymu luciferázy a následně je světelný signál detekován a vyhodnocen programem.
- Luciferáza + D-luciferin + ATP → Luciferáza-luciferin-AMP + PPi
- Luciferáza-luciferin-AMP + PPi + O2 → Luciferáza + Oxyluceferin + AMP + CO2 + světlo
- Luciferáza + D-luciferin + ATP → Luciferáza-luciferin-AMP + PPi
Poslední enzymatickou reakcí je reakce apyrázy, která odstraní nezainkorpované nukleotidy a ATP, aby následně mohlo dojít k zopakování celého výše popsaného procesu a mohlo být analyzováno zařazení dalšího nukleotidu. Tato degradace je nezbytná, aby bylo zajištěna synchronizace mezi syntézou a detekcí světelného signálu.
- ATP→AMP + 2Pi (apyráza)
- dNTP→dNMP + 2 Pi (apyráza)
- ATP→AMP + 2Pi (apyráza)
Aplikace
Při pyrosekvenování je možné sekvenovat nejméně 20 nukleotidů dlouhé úseky. To přináší řadu možností, na co je možné pyrosekvenování využít. Jednou z možností je hledání dosud neidentifikovaných mutací. Hledáme-li mutaci v genu, jehož konsensus sekvence je známá, lze přidávat do pyrosekvenační reakce nukleotidy podle této konsensus sekvence. Tak bude celý proces rychlejší a mutace bude identifikována s velkou přesností. Podobně lze pyrosekvenování využít pro je tzv. Single nucleotide polymorphism (SNP) genotyping, kdy je snaha zjistit všechny možné existující alely daného genu. Dalším použitím je genotypizace bakterií. Pro rozlišení různých bakteriálních kmenů se využívá sekvence 16S rRNA, která obsahuje jak silně konzervované oblasti, tak variabilní oblasti. Na základě sekvence variabilní oblasti lze od sebe odlišit patogenní kmeny od nepatogenních kmenů jednoho bakteriálního druhu. Pyrosekvenování umožňuje tuto analýzu provést rychle a efektivně. Podobně se využívá i genotypizace virů. U viru HIV se určuje touto metodou rezistence vůči používaným léčivům. Bylo využito i pro lidské papilomaviry a virus hepatitidy B a C. Pyrosekvenování lze také využít pro určení tzv. tagů, neboli značek. Předpokládá se, že každý transkript lze identifikovat na základě krátké značky (v délce do 30 nukleotidů). Pomocí pyrosekvenování byla zjištěna celá řada nových značek využitelná pro rychlé určení transkriptomu. [2]
Reference
- ↑ a b AHMADIAN, Afshin, Ehn, Maria; Hober, Sophia. Pyrosequencing: History, biochemistry and future. Clinica Chimica Acta. NaN-NaN-NaN, roč. 363, čís. 1–2, s. 83–94. DOI 10.1016/j.cccn.2005.04.038.
- ↑ AHMADIAN, Afshin; EHN, Maria; HOBER, Sophia. Pyrosequencing: History, biochemistry and future. S. 83–94. Clinica Chimica Acta [online]. Leden 2006. S. 83–94. Dostupné online. DOI 10.1016.
Média použitá na této stránce
Autor: Gharizadeh B, Kalantari M, Garcia C, Johansson B, Nyrén P, Licence: Copyrighted free use
Schematické znázornění automatizovaného pyrosekvenačního systému