Sekvenování DNA
Sekvenování DNA (též sekvenace či sekvencování, mnohdy také „čtení“ DNA) je souhrnný termín pro biochemické metody, jimiž se zjišťuje pořadí nukleových bází (A, C, G, T) v sekvencích DNA.[1] Tyto sekvence jsou součástí dědičné informace v jádru (u prokaryot spíše tzv. nukleoid), dále však i v plazmidech, mitochondriích a plastidech.
Dnes je známo obrovské množství metod sekvenování DNA. Od sedmdesátých let 20. století je používána zejména metoda Fredericka Sangera, která využívá v klasické podobě dideoxynukleotidů a následné elektroforézy. V poslední době se do popředí dostává hlavně pyrosekvenování a další metody, jež slibují zrychlení i zlevnění celého procesu. Sekvenování DNA je užitečné nejen v základním výzkumu biologických procesů, ale i v aplikovaných oborech, jimiž je diagnostika nemocí či forenzní medicína. Sekvenování se uplatnilo např. v projektu čtení lidského genomu (Human Genome Project), ale přečteny byly genomy i mnoha jiných organismů, včetně různých rostlin, živočichů a mikroorganismů. Někdy se sekvenují pouze jisté části genomu, které mají pro výzkumníky v daném okamžiku význam.

Historie výzkumu

Ještě na začátku sedmdesátých let bylo určení pořadí nukleotidů velmi obtížné a obvykle se provádělo nepřímo, tedy sekvenováním RNA molekul či proteinů.[2] První sekvenování krátké sekvence DNA provedl v roce 1970 Ray Wu z Cornellovy univerzity; tato sekvence se však skládala z pouhých 12 nukleových bází a pocházela z okrajových regionů genomu viru lambda. Práce Wuovi a jeho spolupracovníkovi trvala 3 roky.[3]
Průlom nastal v druhé polovině 70. let. V roce 1975 vyvinul svou sekvenační metodu Frederick Sanger a A. R. Coulson.[4] Allan Maxam a Walter Gilbert vyvinuli další, poměrně rychlou metodu sekvenování DNA v roce 1977[4] a druhý jmenovaný dostal za svou vědeckou činnost v roce 1980 Nobelovu cenu.[5] V počátcích sekvenování bylo možno osekvenovat za den jen několik tisíc nukleových bází a sekvenování celého genomu bylo velice náročné. Postupně však byly metody modernizovány a dnes má vědecká veřejnost k dispozici v databázích sekvence DNA čítající několik stovek miliard nukleových bází.[6] Prvním zcela osekvenovaným genomem byl genom bakterie Haemophilus influenzae, dnes však je k dispozici široké rozpětí genomů různých jiných organizmů.[2] V roce 2001 byla zveřejněna první podoba genomu člověka,[7][8] poslední data však byla publikována až v roce 2006.[9]
Principy sekvenování
Při zjišťování pořadí nukleotidů v DNA, popřípadě v RNA, lze uplatnit několik různých principů. Různé metody sekvenování mohou využívat i stejných principů.
- sekvenace štěpením – sekvenace se dosáhne sekvenčně specifickým chemickým nebo biochemickým štěpením vlákna DNA a následnou charakterizaci fragmentů
- sekvenace syntézou – techniky využívající DNA polymerázu a sledující identitu nebo pozici nukleotidů v syntetizovaných fragmentech; pro účely sledování jsou nukleotidy nebo fragmenty značeny buď izotopy nebo fluorescenčně
- sekvenace ligací – podobné sekvenaci syntézou, ale vznik delších fragmentů je zabezpečován aktivitou DNA ligázy
- sekvenace hybridizací – detekce nukleotidů probíhá na základě jejich schopnosti vázat se na (hybridizovat se) sondami (vlákny DNA se známou sekvencí)
- sekvenace na pórech – identifikace nukleotidů probíha na základě změny elektrického proudu procházejícího pórem umístěným v jinak nepropustné membráně, kterým v době měření prochází molekula DNA
- sekvenace mikroskopem – sekvenace spočívá ve vizualizaci vlákna DNA na úrovni zvětšení, kde už lze odlišit od sebe různé báze
Metody sekvenování

Bylo vyvinuto poměrně velké množství technik sloužících k sekvenování DNA, které se liší některými základními principy a dále především cenou a rychlostí. Dvěma základními metodami jsou Maxamovo-Gilbertovo sekvenování a Sangerovo sekvenování.[4][10] Obě mají společné to, že využívají k roztřídění sekvencí gelovou elektroforézu; liší se však způsoby, jak tyto sekvence vznikají. Sangerova metoda je výhodnější zejména v tom, že se při ní do takové míry nemusí využívat toxické látky. Modernizací Sangerovy metody je také možné sekvenování zjednodušit či zrychlit (fluorescenční obarvování atd.).[3]
Maxamova–Gilbertova metoda
Metoda, kterou vyvinuli Allan Maxam a Walter Gilbert, se též označuje jako M&G nebo též „chemické sekvenování“. Vzorek obsahuje krátkou sekvenci DNA (a to jak dvouvláknovou či jednovláknovou), která je na svém 5' konci radioaktivně označena fosforem 32P. Tento vzorek se rozdělí v klasickém případě na pět částí a každá je vystavena chemikáliím, které specificky štípají sekvenci DNA v místě, kde rozpoznají jistou nukleovou bázi. První z pěti nádob například obsahuje dimethylsulfát a piperidin, načež se zahřátím rozštěpí sekvence v místě, kde je přítomen guanin. Druhá nádoba obsahuje hydrazin a piperidin, tyto chemikálie rozštípají DNA v místech, kde je cytosin nebo thymin. Tímto způsobem je v každé z pěti nádob docíleno různě dlouhých sekvencí DNA.[10]
Všechny sekvence DNA se následně umístí vedle sebe do polyakrylamidového gelu a spustí se elektroforéza. Tato metoda způsobí, že se v gelu seřadí všechny sekvence podle své délky: nejdále v gelu doputují nejkratší sekvence. Dalším krokem je přiložení tohoto gelu k filmu citlivému na rentgenové záření. Na vyvolaném filmu jsou pak všechny sekvence z gelu s 5' koncem označeným radioaktivním fosforem patrné jako svítící proužky (metoda označovaná jako autoradiografie[11]). Nakonec se podle pozice těchto proužků ve srovnání s ostatními proužky vyhodnocuje, jaké bylo původní řazení nukleových bází ve vzorku DNA.[10]
Sangerova metoda

Sangerova metoda (též označovaná „metoda plus a minus“, „dideoxy metoda“ či „metoda primed synthesis“) je použitelná k sekvenování krátké sekvence jednovláknové DNA. V podstatě využívá biologického procesu replikace DNA. Vybraná sekvence se vloží do reakční směsi, jež obsahuje vhodný radioaktivně označený primer (α-P32-dATP), DNA polymerázu (Taq či T7 polymeráza), zásobu čtyř esenciálních deoxyribonukleotidů (dATP, dGTP, dCTP, dTTP), ale navíc také jeden ze čtyř dideoxynukleotidů. Dideoxynukleotid je schopen se začlenit do replikující se DNA, ale následně zastaví elongaci řetězce, protože nemá OH skupinu, na níž by se připevnil další nukleotid. Každý dideoxynukleotid se vloží do jedné ze čtyř nádob se vzorkem a všechny replikované sekvence v dané nádobě tedy zákonitě skončí dideoxynukleotidem svého typu.[10]
Výsledkem je směs různě dlouhých sekvencí DNA, které začínají radioaktivním primerem a končí daným dideoxynukleotidem. Když se seřadí na elektroforéze (podobně jako u metody Maxam–Gilbert) podle délky, můžeme snadno porovnáním čtyř vedle sebe umístěných elektroforetických gelů zjistit, jak za sebou následovaly nukleové báze ve zkoumané sekvenci DNA.
Sangerova metoda má mnoho alternativ. Podle jednoho protokolu se do nádob nepřidávají dideoxynukleotidy, nýbrž vždy všechny deoxynukleotidy (dNTP) vyjma jednoho – syntéza dané sekvence skončí tehdy, následuje-li právě ta nukleová báze, která není k dispozici v reakční směsi. „Plus“ metoda zase pracuje v každé nádobě pouze s jedním deoxynukleotidem – sekvence se vytvoří tehdy, je-li na řadě právě tento deoxynukleotid.[4] Revolučním krokem je další metoda, která zase využívá fluorescenční barviva navázaná na jednotlivé dideoxynukleotidy. Tato metoda je však v principu opět podobná Sangerovu původnímu protokolu – prodlužování sekvence skončí, když se naváže jeden z dideoxynukleotidů. Má však výhodu v tom, že je možné místo čtyř zkumavek použít jenom jedinou, protože při elektroforéze jsou jednotlivé nukleové báze označené různými fluorescenčními barvami. Tato tzv. kapilární elektroforéza může být do velké míry automatizována a tedy velice zrychlena.[12]
Pyrosekvenování
Pyrosekvenování (angl. pyrosequencing) je jedna z novějších metod sekvenování DNA. Je sice (podobně jako Sangerova metoda) založena také na syntéze nových sekvencí DNA, ale liší se způsobem, jak je detekováno začlenění daného nukleotidu (nevyžaduje elektroforézu).[13] Pyrosekvenování vyvinul v roce 1996 ve Stockholmu profesor Pål Nyrén se svým studentem Mostafou Ronaghi.[14][15][16] Ve směsi pro pyrosekvenování musí být přítomno velké množství enzymů, mimo DNA polymerázy ještě ATP sulfuryláza, luciferáza a apyráza; ze substrátů pak adenosinfosfosulfát a luciferin. Do této směsi jsou postupně vkládány nukleotidy různých typů (dATP, dGTP, dCTP, dTTP). Když se po přidání jednoho z nich uvolní světelné záření, znamená to, že se do vznikajícího řetězce začlenil jeden nebo více nukleotidů tohoto typu. Toto světlo zde vzniká v důsledku enzymatické reakce, na jejímž začátku je uvolnění pyrofosfátu z nově začleněného nukleotidu a na jejímž konci je spotřeba vzniklého ATP luciferázou k oxidaci luciferinu (viz video na stránkách výrobce).[17] Před přidáním dalšího nukleotidu je původní nukleotid (již obsažený v roztoku) rozložen.
Nejnovější metody
Samozřejmě se neustále objevují nové metody označované anglickým termínem NGS (next-generation sequencing), které slibují zefektivnění, zjednodušení, urychlení či zpřesnění DNA sekvenování, aby bylo schopné přečíst co nejrychleji velké genomy (viz následující kapitola). K těmto dnes již zavedeným metodám patří například:
- 454 sekvenování (též „polony sequencing“) zkomercionalizovala firma 454 Life Sciences. Využívá pyrosekvenování, ale předtím, než k samotnému sekvenování dojde, jsou jednotlivé fragmenty DNA umístěny na streptavidinový substrát a každý z těchto fragmentů je umístěn do speciálního pikolitrového reaktoru. Paralelně probíhá obrovské množství sekvenovacích procesů najednou a výsledek je čten počítačem.[18][19][20]
- SMRT (Single molecule real-time) je metoda, která sleduje v reálném čase probíhající replikaci molekuly DNA, a to ve velmi drobné nádobce o objemu 20 zeptolitrů, kde je umístěno pouze 1 vlákno DNA a jediná DNA polymeráza. Jednotlivé báze jsou fluorescenčně obarvené a když se začlení do prodlužujícího se řetězce DNA, jejich fluorescenční barvivo se uvolní a vydá intenzivní záblesk. Tento záblesk je zachycen detektory a vyhodnocen jako odpovídající nukleotid.[21]
- Technologie nanopórů využívají velmi tenkých otvorů na molekulární úrovni, jimiž prochází zkoumaný řetězec DNA. V nanopóru jsou navíc umístěny drobné elektrody. Tím, jak každý ze čtyř nukleotidů ovlivňuje svým vlastním specifickým způsobem proud elektronů mezi elektrodami, je možné velmi rychle určit pořadí procházejících nukleotidů.[6]
Sekvence získané sekvenováním nejnovějšími metodami (NGS) jsou zpravidla velmi objemné soubory obsahující velké množství párů bazí. Jedním z těchto typů souborů je například textový soubor FASTQ. Pro kontrolu kvality získaných sekvencí se obvykle používá program kontrolující kvalitu sekvencí, jeden z nich je například program FastQC.
Sekvenování genomu
Genomy jsou zpravidla velmi rozsáhlé a jejich sekvenování nukleotid po nukleotidu není ani při použití nejmodernějších postupů otázkou okamžiku. Dalším omezením je cena: sekvenování lidského genomu (Human Genome Project) stálo téměř tři miliardy amerických dolarů,[22] nově patentované technologie se však snaží srazit tuto částku na 100 000 dolarů, v některých případech dokonce na pouhých 1000 dolarů.[6] Běžné sekvenovací metody navíc využívají obvykle separaci pomocí elektroforézy, a tak dokážou přečíst na jeden zátah pouze několik stovek[23] (udává se 300–800[24]) nukleových bází. Existuje však několik strategických metod, jak přečíst toto obrovské množství genetického materiálu:
- „Whole–genome shotgun“ metoda – při této metodě se genom rozštípe na náhodně velké fragmenty, každý z nich se osekvenuje a následně se tyto sekvence seřadí (pomocí počítačového softwaru). Toto seřazení však komplikují různé repetitivní úseky.[23] Navíc je nutné celý genom v podstatě osekvenovat několikrát (asi 7–9krát[24]), aby jednotlivé sekvence dostatečně přesahovaly a bylo možné podle těchto přesahů sestavit genom.
- BAC sekvenování („Clone-by-clone“) – využívá se tzv. BAC plazmidů (BAC – bacterial artificial chromosome). Postup je poněkud opačný, než u předchozí metody. Nejprve se vytvoří jakási mapa celého genomu, jež rozdělí tento genom na sekvence o délce asi 150 000 párů bází, každá jednotlivá sekvence se vloží do bakterie v podobě uměle připraveného plazmidu, načež se jednotlivé klony mohou sekvenovat – a to vlastně shotgunovou metodou v malém měřítku. Výhodou je, že u této metody odpadá často složitá práce se seřazováním obrovského množství sekvencí, jako u předchozí metody.[23]
Rychlost sekvenování
Vhodným měřítkem k porovnávání rychlosti jednotlivých metod sekvenování DNA je lidský genom. Před deseti lety trvalo Celera Genomics a HGP několik let, než přečetli genom člověka. V roce 2008 byl genom Jamese Watsona přečten za pouhých několik měsíců. Nové technologie, které se pomalu dostávají do ruky vědcům, slibují, že bude již brzy možné stejnou práci vykonat v řádu minut.[25] Archon X Prize je ocenění spojené s finanční prémií 10 milionů dolarů, které získá tým, jež bude schopen s dostatečnou přesností osekvenovat 100 lidských genomů během 10 dní, a to za cenu nanejvýš 10 000 dolarů na jeden osekvenovaný genom.[26]
Využití

Sekvenování DNA má množství aplikací ve vědě, medicíně i například v zemědělství. Sekvenují se celé genomy, případně jenom některé části, které mají pro daný obor význam. Například pro studium evolučního vývoje organizmů se zpravidla sekvenuje DNA, jež kóduje ribozomální RNA. V kriminalistice zase mají význam repetitivní oblasti DNA zvané mikrosatelity. Někdy se sekvenují jen ty části genomu, které kódují geny: tato filosofie se nazývá EST (expressed sequence tag).
V lékařství slibuje sekvenování DNA například revoluci v diagnostice chorob a časnému zjištění náchylnosti jedince k určitým nemocem (rakovina, kardiovaskulární onemocnění). Usnadňuje se také výroba léků – vědci mohou zacílit lék proti konkrétnímu produktu konkrétního genu. Podle genotypu jedince může být vybírán správný léčebný postup. Pokud bude zjištěna jistá genetická porucha, nabízí se v budoucnosti rovněž možnost genové terapie.[27]
Ve fylogenetice představuje DNA sekvenování možnost studovat fylogenezi (evoluční vývoj) organizmů. Antropologie zase využívá srovnávání DNA k zjišťování migrací lidských ras (zejména podle mitochondriální DNA a Y-chromozomální DNA). Ve forenzních vědách může osekvenovaná DNA působit jako důkaz něčí viny či neviny v zločinu, dá se pomocí ní určit otcovství a podobně. V zemědělství představují DNA technologie celou plejádu nových možností včetně různých geneticky modifikovaných plodin.[27]
Odkazy
Reference
- ↑ RACLAVSKÝ, Vladislav. Metody molekulární genetiky [online]. Ústav biologie Lékařské fakulty Univerzity Palackého v Olomouci, 2003 [cit. 2009-06-20]. Kapitola 8. Sekvenování DNA. Dostupné v archivu pořízeném dne 2010-02-28.
- ↑ a b ALBERTS, Bruce, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, Peter Walter. Molecular Biology of the Cell. 4. vyd. [s.l.]: GarlandScience; NCBI Dostupné online. Kapitola Isolating, Cloning, and Sequencing DNA.
- ↑ a b Charles R. Cantor, Cassandra L. Smith. Genomics: The Science and Technology Behind the Human Genome Project. [s.l.]: John Wiley & Sons, Inc., 1999. Dostupné online. ISBN 0-471-59908-5.
- ↑ a b c d ROBERT C. KING; WILLIAM D. STANSFIELD; PAMELA K. MULLIGAN. A Dictionary of Genetics, Seventh Edition. [s.l.]: Oxford University Press, 2006.
- ↑ nobelprize.org [online]. [cit. 2009-08-27]. Dostupné v archivu pořízeném dne 2010-12-12.
- ↑ a b c MITCHELSON, Keith R. New high throughput technologies for DNA sequencing and genomics. [s.l.]: [s.n.], 2007. Dostupné online. S. 381.
- ↑ McPherson JD, Marra M, Hillier L, et al.. A physical map of the human genome. Nature. February 2001, roč. 409, čís. 6822, s. 934–941. DOI 10.1038/35057157. PMID 11237014.
- ↑ Venter JC, Adams MD, Myers EW, et al.. The sequence of the human genome. Science (journal). February 2001, roč. 291, čís. 5507, s. 1304–1351. DOI 10.1126/science.1058040. PMID 11181995.
- ↑ Gregory SG, Barlow KF, McLay KE, et al.. The DNA sequence and biological annotation of human chromosome 1. Nature. May 2006, roč. 441, čís. 7091, s. 315–321. DOI 10.1038/nature04727. PMID 16710414.
- ↑ a b c d RÉDEI, George P. Encyclopedia of Genetics, Genomics, Proteomics, and Informatics. 3rd Edition. vyd. [s.l.]: Springer, 2008. ISBN 978-1-4020-6753-2.
- ↑ Stenesh, J. (1989): Dictionary of Biochemistry and Molecular Biology (2nd Edition). John Wiley & Sons.
- ↑ Archivovaná kopie. apendix.bf.jcu.cz [online]. [cit. 2009-09-03]. Dostupné v archivu pořízeném dne 2009-02-28.
- ↑ Definition of pyrosequencing from the Nature Reviews Genetics Glossary [online]. [cit. 2008-10-28]. Dostupné online.
- ↑ Ronaghi et al. A sequencing method based on real-time pyrophosphate. Science. 1998-07-17, roč. 281, s. 363. Dostupné online. DOI 10.1126/science.281.5375.363. PMID 9705713.
- ↑ Ronaghi et al. Real-time DNA sequencing using detection of pyrophosphate release. Analytical Biochemistry. 1996, roč. 242, s. 84–89. DOI 10.1006/abio.1996.0432. PMID 8923969.
- ↑ Nyrén, P. The History of Pyrosequencing. Methods Mol Biology. 2007, roč. 373, s. 1–14. PMID 17185753.
- ↑ Principle of Pyrosequencing Technology [online]. Qiagen [cit. 2009-09-01]. Dostupné v archivu pořízeném dne 2009-09-05.
- ↑ Margulies M, Egholm M, Altman WE, et al. Genome sequencing in microfabricated high-density picolitre reactors. Nature. September 2005, roč. 437, čís. 7057, s. 376–80. DOI 10.1038/nature03959. PMID 16056220.
- ↑ Shendure, J. Accurate Multiplex Polony Sequencing of an Evolved Bacterial Genome. Science. 2005, roč. 309, s. 1728. DOI 10.1126/science.1117389.
- ↑ Applied Biosystems' SOLiD technology. solid.appliedbiosystems.com [online]. [cit. 04-09-2009]. Dostupné v archivu pořízeném dne 16-05-2008.
- ↑ SMRT™ Technology At a Glance [online]. Pacific Biosciences. Dostupné online.
- ↑ http://www.genome.gov/11006943
- ↑ a b c ALBERTS, Bruce, et al. Essential Cell Biology. 2. vyd. New York: Garland Science, 2004. Dostupné online.
- ↑ a b Genomika [online]. Katedra fyziologie Přírodovědné fakulty UK v Praze. Dostupné online.[nedostupný zdroj]
- ↑ The 15-Minute Genome 2009 Industrial Physics Forum features faster, cheaper genome sequencing (tisková zpráva) [online]. 2009. Dostupné online.[nedostupný zdroj]
- ↑ "PRIZE Overview: Archon X PRIZE for Genomics". genomics.xprize.org [online]. [cit. 2009-09-04]. Dostupné v archivu pořízeném z originálu.
- ↑ a b DNA sequencing “the technology lecture” [online]. George Mason University; Department of Bioinformatics and Computational Biology [cit. 2009-09-04]. Dostupné v archivu pořízeném dne 2010-06-14.
Související články
- Sekvenátor
Externí odkazy
 Obrázky, zvuky či videa k tématu sekvenování DNA na Wikimedia Commons
Obrázky, zvuky či videa k tématu sekvenování DNA na Wikimedia Commons
Média použitá na této stránce
Example of a pyrogram as displayed after a pyrosequencing session. Produced by Zaimon.
Electrophoresis of short sequences created by Maxam-Gilbert sequencing technique. The primary sequence, GTCATAGCA, is read from the bottom of the polyacrylamide gel
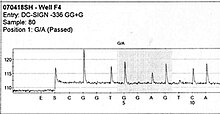
Výsledek sekvenace úseku DNA automatickým sekvenátorem
Two arrows symbolising an article being split in two.
Autor: John Schmidt, Licence: CC BY-SA 3.0
DNA sequencing gel. Sequence visualized by autoradiography.
Autor: Vojtech.dostal, Licence: CC BY-SA 3.0
Phylogenetic tree of Eukaryota. Three groups in the base of the tree are incertae sedis, all other groups fall into one of the five 'kingdoms' (brown coloured). Animals, the old-style 'kingdom Animalia', are shown as 'metazoa' in this chart. For more details, see this article (Simpson et Roger 2004)






