RNA

Ribonukleová kyselina (RNA, česky dříve RNK) je nukleová kyselina tvořená vláknem ribonukleotidů, které obsahují cukr ribózu a nukleové báze adenin, guanin, cytosin a uracil. Je zodpovědná za přenos informace z úrovně nukleových kyselin do proteinů a u některých virů je dokonce samotnou nositelkou genetické informace. V mnoha ohledech je podobná deoxyribonukleové kyselině (DNA), od které se liší jednak přítomností ribózy, kterou má ve své cukr-fosfátové kostře namísto deoxyribózy, a také tím, že využívá nukleovou bázi uracil namísto thyminu. Díky větší reaktivitě ribózy může molekula RNA zaujímat větší množství prostorových uspořádání a zastávat mnohem více funkcí, než mnohem stabilnější DNA, která je využívaná buňkou především jako úložiště genetické informace. Molekula RNA je také na rozdíl od DNA obvykle jednovláknová, často ovšem díky vnitřnímu párování zaujímá složitější strukturu, a v některých případech, například u některých virů, se vyskytuje i dvouvláknová RNA.
RNA má v těle řadu funkcí, z nichž hlavní je zajištění překladu genetického kódu, tedy převod informace z DNA do struktury proteinů. Oblast DNA nesoucí gen je nejprve přepsána (procesem transkripce) do mediátorové RNA. Ta je následně přeložena (procesem translace) do proteinů tvořených řetězcem aminokyselin. Zařazení správné aminokyseliny při tvorbě proteinů zajišťuje vazba transferové RNA na specifický kodón v mRNA pomocí párování jejich bází. Samotný překlad genetického kódu probíhá na ribozomu, který je složený z RNA (tzv. rRNA) i proteinů, přičemž RNA v ribozomu tvoří nejen strukturní složku, ale je zodpovědná i za syntézu peptidové vazby v nově vznikajícím proteinu. Ribozom je tedy významným zástupcem skupiny RNA s katalytickou aktivitou, tzv. ribozymů.
Kromě už zmíněných rolí v překladu genetického kódu nebo strukturních a katalytických funkcí, hraje RNA roli v řadě dalších buněčných pochodů, jako je úprava RNA, jemná kontrola translace pomocí RNA interference, funguje jako templát pro syntézu telomer, atd. Podle hypotézy RNA světa mohla být RNA pro svou všestrannost, schopnost nést i replikovat genetickou informaci a syntetizovat podle ní proteiny první nukleovou kyselinou využívanou živými organismy.
Historie výzkumu RNA

RNA byla poprvé izolována roku 1868 ve směsi s DNA F. Miescherem[1] a nazvána nuklein, nuklein byl ale původně považován za pouhý buněčný sklad fosfátu. Existenci dvou různých nukleových kyselin naznačovaly výzkumy Albrechta Kossela, který mezi lety 1885–1901 popsal všechny hlavní báze a zjistil, že nukleové kyseliny obsahují cukry, které charakterizoval jako pentózy.[2] Až do 30. let 20. století panovala představa, že živočichové mají v jádře DNA, která byla podle izolace z thymu nazvána „thymovou nukleovou kyselinou“ a nižší eukaryota a rostliny obsahují RNA, která byla nazývána „kvasinkovou nukleovou kyselinou“.[2] Chemickou strukturu nukleových kyselin upřesnil Phoebus Levene, který roku 1909 zjistil, že RNA obsahuje ribózu a roku 1929, že DNA obsahuje deoxyribózu,[2] z čehož vychází současné pojmenování těchto nukleových kyselin. Levene také objevil způsob, jakým jsou jednotlivé složky nukleových kyselin propojeny – objevil cukr-fosfátovou kostru a navrhl tzv. tetranukleotidovou hypotézu, podle které jsou nukleové kyseliny tvořené pouhou čtveřicí nukleotidů.[3] Levenova tetranukleotidová hypotéza (publikována 1919) nesprávně naznačovala, že nukleové kyseliny jsou příliš jednoduché na to, aby mohly nést genetickou informaci, a výzkum RNA a nukleových kyselin se na dlouhou dobu zastavil.[2]
Role RNA v syntéze proteinů byla naznačována už v roce 1939, protože množství DNA v nedělící se buňce zůstává konstantní, ale množství RNA se zvyšuje u aktivních buněk a snižuje se u neaktivních, hladovějících;[4] u aktivních buněk se také vyskytují výrazná ribonukleoproteinová tělíska,[5] později pojmenovaná jako ribozomy. Existenci mRNA předpovídal Francis Crick ve svém centrálním dogmatu molekulární biologie (1958[6]), protože očekával, že mezi DNA a proteiny musí být nějaký prostředník. Crick spolu s řadou dalších vědců založili RNA-tie club („klub RNA kravat“), který si dal za cíl odhalit genetický kód. Členové RNA-tie klubu v 50. letech 20. století popsali kodón, teoreticky vypočetli, že počet nukleotidů v kodónu je právě tři, a postulovali adaptérovou hypotézu předpovídající existenci tRNA. Předpovězené molekuly mRNA[7] a tRNA[8] byly později skutečně objeveny koncem 50. let. Tou dobou se už také dlouho předpokládáno, že syntéza proteinů probíhá na ribozomech, což bylo potvrzeno roku 1961.[9]

Dalším klíčovým momentem výzkumu RNA byla izolace enzymu polynukleotid fosforylázy, který je schopen za určitých podmínek syntetizovat RNA, za což získal objevitel Severo Ochoa roku 1959 Nobelovu cenu za medicínu.[10] I když bylo později zjištěno, že tento enzym přirozeně slouží k degradaci RNA a skutečná RNA polymeráza byla objevena až v době udělení této Nobelovy ceny,[11] syntéza RNA umožnila rozluštit genetický kód: prvním zjištěním bylo, že RNA tvořená výhradně spojením uridinu (polyuracil) dává vzniknout polypeptidu tvořenému pouze molekulami fenylalaninu, což znamená, že kodón „UUU“ kóduje aminokyselinu fenylalanin.[9] Zbytek genetického kódu už byl rozluštěn během několika let a výzkum byl roku 1968 oceněn Nobelovou cenou.[12] Po popsání klíčové role RNA v syntéze proteinů následovala velká vlna zájmu o RNA související s výzkumem genetického kódu a molekuly RNA účastnící se zpracování genetického kódu byly detailně zkoumány.[13] tRNA byla první osekvenovanou (přečtenou) nukleovou kyselinou[13] a první nukleovou kyselinou s určenou krystalickou strukturou.[14][15] Díky pokrokům v analýze RNA byl také přečten první genom – RNA genom bakteriofága MS2.[16]
Postupně začalo být jasné, že RNA má i jiné funkce, než je zpracování genetického kódu, a roku 1967 Carl Woese navrhl hypotézu RNA světa,[17] podle které byly molekuly RNA původní nositelkou genetické informace a zároveň zajišťovaly potřebné katalytické funkce. V 70. letech byla tato teorie podpořena popsáním reverzní transkriptázy, která slouží k přepisu RNA do DNA retrovirům,[18] a v 80. letech objevem katalyticky aktivních RNA, tzv. ribozymů: bylo zjištěno, že v ribozomu[19] a RNáze P[20] (zodpovědné za zrání tRNA) je v katalytickém centru právě molekula RNA.
Během toho pokračoval další výzkum a byly objeveny nekódující RNA, ribozomální RNA sdílená všemi živými organismy začala být využívána pro určení evoluční příbuznosti, bylo zjištěno, že RNA je po své syntéze složitě upravována, například sestřihem nekódujících intronů, polyadenylací nebo například editací a regulována procesem RNA interference. Výzkum RNA pomohl odhalit podstatu celé řady klíčových biologických procesů a za studium RNA byla Nobelova cena udělena více než 30 lidem.
V současnosti probíhá bouřlivý výzkum na mnoha polích biologie RNA. Bádá se například v oblasti propojení molekul RNA a epigenetických regulací či na poli RNA interference. Projekt ENCODE zase odhalil, že zatímco pouze asi 1,5 % lidského genomu přímo kóduje proteiny, tak až 80 % genomu může být přepisována do podoby RNA. Některé léky využívané v léčbě jsou založeny na RNA, ve fázi klinického testování je přes 50 takových potenciální léků.[21] Schválen byl zatím pouze RNA aptamer Pegaptanib využívaný pro léčbu některých typů makulární degenerace, a to roku 2004 v USA a v EU o rok později,[22][23] a léky založené na antisense RNA: Fomivirsen, antivirotikum proti lidskému cytomegaloviru,[24] a Mipomersen proti familiární hypercholesterolemii.[25] Zajímavé se zdá především terapeutické využití RNA interference, která představuje alternativu ke klasické genové terapii, všechny potenciální léky tohoto typu jsou ale zatím ve fázi klinického testování.[26]
Stavba

Chemická struktura
Ribonukleová kyselina je biopolymer tvořený ribonukleotidy, což jsou látky složené z nukleové báze (adenin (A), guanin (G), cytosin (C) nebo uracil (U)), pětiuhlíkatého monosacharidu ribózy a jednoho zbytku kyseliny fosforečné. Samotná nukleová báze napojená na ribózu se nazývá ribonukleosid, až jeho fosforylovaná forma nacházející se v RNA je ribonukleotid. V kostře RNA se střídá ribóza s fosfátovou skupinou, které jsou vzájemně kovalentně propojené fosfodiesterovou vazbou. Na ribózu jsou v pozici 1' připojeny glykosidickou vazbou nukleové báze, které představují vlastní jednotky dědičnosti a právě pomocí nich je v RNA zapsána informace. První tři báze jsou totožné s těmi, které se nacházejí v DNA, ale thymin přítomný v DNA je nahrazen uracilem, který se od thyminu liší nepřítomností methylové skupiny.
Dalším významným rozdílem je to, že ribóza nesená v RNA má oproti deoxyribóze tvořící DNA na pozici 2' hydroxylovou (OH) skupinu. RNA je kvůli volné 2' OH skupině výrazně reaktivnější a flexibilnější, ale také nestabilnější a v zásaditém prostředí dochází k hydrolýze fosfodiesterové vazby blízkou -OH skupinou, což vyvolává rozštěpení kostry RNA.[27] Kromě toho je RNA citlivější k degradaci řadou enzymů. Reaktivita RNA jí ovšem umožňuje v mnohých případech působit jako katalyzátor chemických reakcí – takovým molekulám RNA se říká ribozymy.[1]
V porovnání s DNA, která vytváří v drtivé většině případů tzv. dvoušroubovici (složenou ze dvou komplementárních vláken), vytváří RNA většinou kratší jednoduchá vlákna, která jsou obvykle jednovláknová. Vyskytuje se i dvouvláknová RNA, která zaujímá dvoušroubovici, pro niž je typické prostorové uspořádání A-formy. RNA je ovšem díky své flexibilitě schopna vytvářet bohaté sekundární a terciární struktury, jejichž variabilita je v porovnání s DNA výrazně bohatší.
Modifikace RNA v buňkách

Do RNA jsou při translaci vneseny pouze čtyři základní báze, následnou modifikací báze nebo ribózy ale vzniká celá řada dalších ribonukleotidů, což rozšiřuje variabilitu molekul RNA. V současné době je známo více než 100 modifikovaných ribonukleotidů, přičemž nejčastější modifikací je 2'-O-methylace ribózy, která je nezbytná pro funkci ribozomu a spliceosomu,[28] a nejvíce různých modifikací se dá nalézt v molekule tRNA.[29] Mezi nejčastější příklady nestandardních složek RNA[pozn. 1] patří dihydrouridin (D), který má destabilizační efekt na strukturu RNA, pseudouridin (Ψ), což je uridin připojený na ribózu neobvykle na své 5' pozici (viz obrázek), jeho role je naopak stabilizace struktury RNA díky své schopnosti tvořit navíc další vodíkový můstek a silnější patrové interakce. Inosin (I) v tRNA umožňuje i nestandardní, tzv. kolísavé párování bází a někdy je využíván ke změně sekvence mRNA při procesu editace RNA.[30] V RNA se dále často vyskytuje 5-methylcytidin (m5C), který v tRNA ovlivňuje funkčnost a v rRNA zvyšuje přesnost translace, jeho funkce v RNA ale není příliš prozkoumaná[31] (naopak její výskyt v DNA je významný epigenetický znak). I když thymin nebývá do RNA zařazen přímo při její syntéze, vyskytuje se velmi často v takzvaném TΨC rameni tRNA, v případě RNA se ale tento nukleosid kvůli jednoznačnosti nazývá ribothymidin nebo 5-methyluridin a ne thymidin. Další významnou modifikací je 7-methylguanosin (m7G), což je nukleosid tvořící neobvyklou strukturu 5' čepičky, která chrání 5' konec eukaryotních mRNA, 7-methylguanosin je navíc připojen k následujícímu ribonukleotidu neobvyklou 5′ – 5′ trifosfátovou vazbou.
Strukturní motivy v RNA
Základní strukturní motivy
Funkce RNA není určena pouze pořadím jejích bází, ale podobně jako u proteinů i její strukturou. RNA dokáže pomocí párování bází a patrových interakcí v rámci svého řetězce vytvářet stejné typy struktur jako DNA, díky flexibilitě kolem své glykosidické vazby a schopnosti vytvářet další vodíkový můstek ale může zaujímat i mnohem složitější uspořádání.
Mezi nejčastější typy prostorového uspořádání RNA patří:
- dvoušroubovice, v případě RNA ovšem pouze tzv. A-formy, která je ve srovnání s B-formou běžnou v DNA širší, má více plochý tvar, hlubší velký žlábek a méně hluboký malý žlábek.
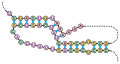
- vlásenka
- vyboulená smyčka vznikající ve dvoušroubovici při lokálním nedokonalém párování bází
- pseudouzel vytvářející několik interakcí v rámci jedné molekuly a kissing loops (tj. „líbající se smyčky“) vznikající mezi různými molekulami
- trojšroubovice, ve které se do klasické dvoušroubovice připojí další vlákno, nejčastěji do malého žlábku
- G-kvadruplex, podobně jako v případě DNA
- Hlavní typy struktur v RNA
- Dvouvláknová RNA tvořená polyuracilem a polyadeninem. (PDBID:1H1K)
- Příklad vlásenky
- Schematické zobrazení pseudouzlu
- Způsob vzniku pseudouzlu
- Reálný příklad pseudouzlu, dvě různá zobrazení
- Schematické znázornění struktury kissing loops
Složitější struktury

Složitější typy struktur se vytvářejí jak pomocí párování bází mezi jednotlivými částmi molekuly, tak i využíváním dalších interakcí, například patrových interakcí mezi ribonukleotidy v různých částech molekuly (tzv. koaxiální skládání) nebo využívání kovových iontů pro stabilizaci řetězce. RNA je také schopná, v mnohem větším rozsahu než DNA, využívat nestandardní typy párování bází, jako je hoogsteenovské párování nebo kolísavé párování bází.[1] Kromě uspořádaných oblastí mají pro funkci RNA význam i oblasti bez stabilní struktury, podobně jako u neuspořádaných oblastí proteinů. Neuspořádané oblasti jsou často místem vazby RNA-vazebných proteinů nebo jiných RNA (například při vazbě mezi tRNA a mRNA) a jsou preferovaným cílem RNA interference.[32] První molekulou RNA se zjištěným prostorovým uspořádáním byla tRNA,[15] a za určení přesné prostorové struktury ribozomu byla roku 2009 udělena Nobelova cena za chemii.[33]
Funkce
Ribonukleová kyselina plní v buňkách mnoho možných úloh. Může například nést informaci o stavbě proteinů, jako je tomu v případě mRNA. Většina RNA přítomná v buňce (kolem 98 %[34]) je ovšem tzv. nekódující a má v buňce naprosto odlišnou roli. RNA může také, podobně jako bílkovinné enzymy, plnit katalytickou (často autokatalytickou) funkci, taková enzymaticky fungující RNA se nazývá ribozym. Pro RNA viry slouží RNA, ať už jednovláknová nebo dvouvláknová, pro uložení vlastní genetické informace.
Čtení genetického kódu

Informace potřebné pro tvorbu proteinů živými organismy jsou v buňkách uloženy na DNA ve formě genů, které kódují molekulu mRNA sloužící jako prostředník. Proces překladu mRNA do proteinů probíhá podle souboru pravidel nazývaných genetický kód, která jsou z velké části uváděna do praxe právě molekulami RNA:
Mediátorová RNA (mRNA, z angl. messenger RNA) je přepisována podle sekvence DNA (u eukaryot je pak exportována do cytoplazmy) a využita pro překlad do proteinů. mRNA je nejdříve přepisována jako prekurzor (hnRNA), který často obsahuje nekódující oblasti, introny. Zralá mRNA stále obsahuje nepřekládané oblasti s regulačními funkcemi a u eukaryot je na koncích opatřena čepičkou a poly(A) koncem, které dále určující její osud v buňce. Samotná kódující oblast nese kodóny tvořené nepřekrývajícími se trojicemi nukleotidů, podle kterých jsou připojeny příslušné aminokyseliny, a start kodon a stop kodon určující začátek a konec transkripce. Aktivně překládaná mRNA je u eukaryot pomocí proteinů rozpoznávajících poly(A) konec a čepičku spojena do pseudokružnice, což umožňuje ribozomům po ukončení translace a jejich odpadnutí rychle znovu nalézt začátek transkripce. Takovou mRNA navíc často překládá více ribozomů najednou a společně tvoří útvar zvaný polyzom.
tRNA (transferová RNA) připojuje specifickou aminokyselinu do rostoucího polypeptidového řetězce při translaci. Slouží jako adaptér pro nabitou aminokyselinu a umožňuje překlad informace z úrovně nukleových kyselin do úrovně proteinů, přičemž samotné rozpoznání probíhá podle párování bází v dvojici kodon-antikodon. V molekule tRNA je průměrně 17 % bází modifikováno,[35] což mimo jiné umožňuje kolísavé párování bází, tedy i jiné než klasické Watson-crickovské párování. Jedna tRNA je proto často schopna rozpoznávat více různých kodónů patřících jedné aminokyselině, což je příčinou degenerovanosti genetického kódu.
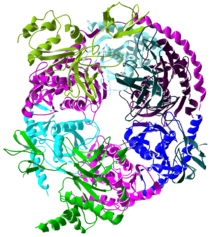
rRNA (ribozomální RNA) je nejčastější molekulou RNA v buňce, u bakterií může tvořit 95–98 % celkové RNA.[36] rRNA je významná jednak svou stavební funkcí v ribozomu, který je tvořen z velké části právě rRNA, jednak tím, že rRNA je zodpovědná za katalytickou aktivitu ribozomu.[19] Důvodů, proč má v ribozomu katalytickou aktivitu RNA a ne protein, může být několik, podle evolučního vysvětlení (tzv. hypotézy RNA světa) byly první proteiny syntetizovány výhradně ribonukleovou kyselinou (bez pomoci proteinů, které se připojily až později) a rRNA už nebyla nikdy nahrazena. Biochemicky se tento fenomén dá vysvětlit tak, že RNA snadno mění konformaci a umožňuje tak nezbytný pohyb ribozomu; navíc právě RNA je nejvhodnější pro specifické rozpoznání dalších RNA řídících syntézu RNA (mRNA a tRNA) pomocí párování bází a dalších interakcí typických pro nukleové kyseliny.[19]
Kromě těchto RNA pro proteosyntézu nezbytných se vyskytují další RNA, které ji přímo regulují. Příkladem je transferová-mediátorová RNA (tmRNA), která u řady bakterií a v plastidech[37] slouží k záchraně „zaseknutého“ ribozomu, k němuž se naváže jako tRNA a je jím přepsána do podoby značky pro degradaci vznikajícího proteinu, což ribozom uvolní.[38] Dalším příkladem je 7SL RNA vyskytující se v signál rozpoznávající částici, 7SL RNA pravděpodobně slouží k zastavení proteosyntézy v průběhu tzv. kotranslační translokace proteinů určených k produkci ven z buňky.[39]
Regulace genové exprese


Kromě toho, že RNA zajišťuje čtení genetického kódu, je také schopná tento proces i dále regulovat. U eukaryot je nejvýznamnějším mechanismem takzvaná RNA interference probíhající na úrovni posttranskripční regulace. Při RNA interferenci je rozštěpena dvouvláknová molekula RNA a jedno z jejích vláken je vneseno do komplexu RISC. Pokud se v buňce vyskytne molekula RNA plně komplementární s vláknem RNA neseném v komplexu RISC, je tato molekula rozštěpena komplexem RISC, pokud je ale druhé vlákno komplementární pouze částečně, mRNA není degradována, ale je zabráněno její translaci.
RNA interferenci řídí dva hlavní typy RNA: siRNA (short interfering RNA) a miRNA (microRNA). siRNA vzniká z dvouvláknové RNA především vnějšího původu (většinou dsRNA viry). Protože je v tomto případě do RISC vneseno jedno z vláken dsRNA, druhé z vláken je tedy plně komplementární a cizorodá dsRNA je proto komplexem RISC rozštěpena. miRNA je naopak kódována buňkou, ať už samostatnými geny pro miRNA, oblastmi uvnitř intronů genů kódujících protein nebo geny pro jinou nekódující RNA. miRNA reguluje genovou expresi tak, že zabraňuje translaci těch mRNA, se kterými nedokonale páruje (příklad viz obrázek). Tento mechanismus se nazývá RNA silencing. Podle bioinformatických analýz se zdá, že až 60 % lidských genů může být regulováno pomocí miRNA[40] a probíhá intenzivní výzkum propojení miRNA a různých nemocí, například rakoviny.[41]
Kromě zmíněných dvou hlavních skupin se rozlišují i další typy RNA řídících RNA interferenci, jako jsou piRNA hrající roli v obraně proti retrotranspozonům a siRNA pocházející z repetitivních sekvencí (rasiRNA). Pro výzkumné nebo léčebné účely se používá umělá shRNA (small hairpin RNA), což jsou krátké molekuly RNA tvořící vlásenku, která je rozeznána a dále zpracována aparátem RNA interference stejně jako prekurzory siRNA nebo miRNA.[42] I když je RNA interference známá pouze z eukaryot, řada bakterií a archeí má pro obranu proti bakteriofágům a dalším parazitickým DNA elementům vyvinut funkční analog RNA interference zvaný CRISPR systém.[43]
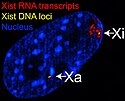
Regulace genové exprese pomocí RNA ale probíhá na mnoha úrovních, například ovlivňováním uspořádání genomu v jádře. Typickým příkladem je dlouhá nekódující RNA nazvaná XIST, která u samic savců inaktivuje jeden z dvou chromozomů X tím, že jej obalí a zabrání aktivaci genů, které tento chromozom nese.
Katalytická funkce

Katalytickou funkci mají v živých organismech především proteinové enzymy, roste ale množství známých molekul RNA, které mají také katalytickou aktivitu, jedná se o tzv. ribozymy. Nejvýznamnějším zástupcem ribozymů je ribozom katalyzující peptidyltransferázovou reakci při vzniku proteinů.[19] Nejčastější reakcí, kterou ribozymy katalyzují, je ovšem štěpení cukr-fosfátové kostry ve vlastní molekule, tyto ribozymy ale de facto nejsou katalyzátory, protože se v průběhu reakce spotřebují. Zajímavým příkladem ribozymů jsou uměle připravené peptidyltransferázy schopné katalyzovat tvorbu vazby mezi aminokyselinami[45] a umělé ribozymy schopné replikovat jiné molekuly RNA,[46] což slouží jako podpora hypotézy RNA světa, podle které byla pro živé organismy první nositelka genetické informace právě RNA. Mezi dobře prozkoumané ribozymy patří RNáza P zodpovědná za zrání tRNA a sebevystřihující introny, což jsou introny (nekódující oblasti) vyskytující se v některých mRNA, tRNA nebo rRNA schopné vyštěpit sebe sama i bez pomoci proteinů. Ribozymy jsou relativně běžné u viroidů a virusoidů, kterým ribozymy štěpí lineární molekulu RNA nesoucí několik násobků jejich genomu, která vzniká při jejich replikaci. V současnosti probíhá klinické testování některých umělých ribozymů, které jsou potenciálně využitelné v léčbě rakoviny[47] nebo virových onemocnění, jako je např. HIV.[48]
Řízení modifikace RNA
Význam RNA pro buňku ilustruje také schopnost RNA modifikovat nebo řídit modifikaci jiných molekul RNA během jejich zrání. V rámci RNA zodpovědných za modifikace RNA je možno vyčlenit několik hlavních tříd nekódujících RNA:
- snRNA (small nuclear RNA, „malá jaderná RNA“) je skupina RNA vyskytující se v jádře eukaryotních buněk, která se podílí na sestřihu prekurzorů mRNA v průběhu jejich zrání. snRNA vytváří ribonukleoproteinové komplexy zvané spliceosom složené ze šesti různých molekul snRNA a 60–150 různých proteinů. snRNA v katalytickém místě spliceozomu je velmi podobná sebevystřihujícím intronům typu II a předpokládá se, že právě z nich spliceosom vznikl, v současnosti ale není jasné, jestli se jedná o ribozym, nebo má katalytickou aktivitu některý z proteinů.[49]
- snoRNA (small nucleolar RNA, „malá jadérková RNA“), vyskytující se v jadérku (často se proto uvádí jako podtyp snRNA), řídí methylaci a pseudouridinylaci rRNA. Do takzvané H/ACA box rodiny snoRNA patří i RNA komponenta lidské telomerázy, která slouží jako templát pro syntézu DNA v oblasti telomer procesem reverzní transkripce.[50]
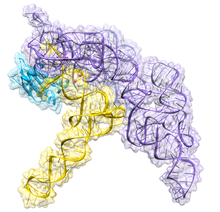
- RNáza P je ribozym nezbytný pro jeden z kroků maturace tRNA. Vyskytuje se v podobě ribonukleoproteinu u bakterií, archeí i eukaryot, u všech skupin organismů je ale RNA katalyzátorem štěpení.[20]
- gRNA (guide RNA) slouží k editaci RNA, což je proces posttranskripční modifikace mRNA, která probíhá v kinetoplastech bičivek (k zástupcům patří například trypanozomy). Při tomto typu editace dochází k přidání nebo odstranění uracilů z molekuly mRNA podle sekvence gRNA.
Vazba malých molekul
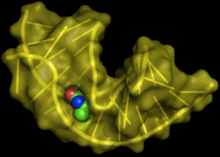
Schopnost RNA tvořit složité struktury jí umožňuje specificky vázat jiné molekuly, takové RNA se nazývají aptamery. V přírodě jsou aptamery součástí tzv. riboswitch („RNA přepínač“), které jsou známé především z bakterií, ale vyskytují se i u rostlin a kvasinek. Riboswitch slouží jako přepínač na molekule mRNA, jejíž konformace se mění v závislosti na tom, jestli je v aptameru navázaná cílová molekula. Typickým příkladem je lyzinový riboswitch nacházející se v řadě bakteriálních mRNA regulujících metabolismus lyzinu. Vazba lyzinu do struktury tvořící riboswitch signalizuje dostatek lyzinu a blokuje překlad těchto mRNA.[51] Aptamery je možné v laboratoři připravit metodou „evoluce ve zkumavce“ (SELEX), při které se z náhodné sady molekul RNA vyberou ty, které jsou schopny vázat požadovanou molekulu, ty se vyizolují, namnoží pomocí nepřesně kopírujících enzymů a proces se opakuje, některé takto získané RNA aptamery se v současnosti využívají jako léky.[21]
Některé ribozymy jsou schopny vázat malé molekuly, které jim slouží jako kofaktory, podobně, jako je tomu u proteinových enzymů.[52] Ribozymy samotné jsou schopny katalyzovat jenom relativně omezený počet chemických reakcí, schopnost RNA vázat také jiné molekuly ale jejich schopnosti rozšiřuje.[53]
Dlouhé nekódující RNA

Dlouhé nekódující RNA (lncRNA) jsou nekódující RNA s délkou alespoň 200 nukleotidů.[54] Projekt ENCODE odhaduje, že jsou v lidském genomu zastoupeny v podobném počtu, jako geny kódující proteiny[55] a představují nejrozšířenější typ nekódující RNA.[56] Tento typ RNA je většinou modifikován podobně, jako mRNA, nese tedy 5' čepičku, je polyadenylován a často sestřižen. Dlouhé nekódující RNA se vyskytují především v buněčném jádře,[57] kde řídí tvorbu jaderných tělísek. U těchto tělísek je běžné, že se jejich jednotlivé komponenty vyskytují v malé koncentraci v celém jádře a shlukují se až tehdy, pokud buňka vyžaduje jejich větší aktivitu, v takovém případě jejich skládání většinou řídí právě dlouhé nekódující RNA.[58][59] Jedná se o Cajalova tělíska, která obsahují RNA specifické pro Cajalova tělíska (scaRNA), které řídí modifikaci malých jaderných RNA (snRNA) a malých jadérkových RNA (snoRNA),[58] „jaderné skvrny“ obsahující NEAT2 řídící úpravu malých jaderných ribonukleoproteinů, ze kterých se skládá funkční spliceosom,[59] a „paraspeckles“, jež jsou centrem RNA editace adenosin→inositol, které jsou vytvářeny kolem dlouhé nekódující RNA NEAT1[59]
Kromě toho dlouhé nekódující RNA ovlivňují přepis genetické informace tím, že regulují epigenetické modifikace v řídících oblastech genů.[60] Významná je především interakce s komplexem polycomb, který je zodpovědný za epigenetické umlčení řady genů pomocí methylace histonů a provází inaktivaci chromozomu X. Asi pětina všech dlouhých nekódujících RNA interaguje s tímto komplexem a předpokládá se, že jsou nějakým způsobem zodpovědné za výběr genu, jehož přepis má být umlčený.[59] Jedním z nejlépe prostudovaných zástupců této třídy RNA je XIST vyvolávající u samic savců inaktivaci chromozomu X tím, že obalí jeden ze dvou chromozomů X a zastaví čtení jeho genetické informace; jako jedna z mála přepisovaných oblastí zůstává okolí gen XIST.
Sobecká RNA
Některé molekuly RNA je možné vnímat jako molekulární parazity. Jsou totiž přítomny v organismu hostitele, někdy jsou dokonce kódovány jeho vlastním genomem, a často se množí na jeho úkor. Jedná se o RNA viry a retroviry, retrotranspozony a sebevystřihující introny. V organismu často zabírají významnou část hostitelského genomu, u člověka zabírá nekódující DNA vzniklá činností těchto elementů téměř polovinu jeho genomu.[61]
RNA viry v užším slova smyslu jsou zástupci virů, kterým RNA slouží jako nositelka genetické informace (viry způsobující chřipku, klíšťovou encefalitidu, SARS,…). Retroviry (např. HIV) mají ve svém genomu RNA, ale využívají zpětný přepis RNA→DNA, jsou schopny svůj genom vložit do genomu hostitele a u člověka tvoří jejich pozůstatky 5–8 % genomu.[61] Retroviry mohou ztratit schopnost se šířit mezi hostiteli a stát se endogenními retroviry. Lidské endogenní retroviry jsou téměř neaktivní, jsou pravděpodobně schopny se šířit pouze mezi pohlavními buňkami svého hostitele.[62] Zajímavý případ lidského endogenního retroviru je HERV-W kódující protein syncytin; tento protein původně sloužil viru pro vstup do buněk, lidský organismus jej ale převzal a využívá jej pro fúzování buněk (trofoblastů) v placentě.[63] Ještě jednodušší než endogenní retroviry jsou retrotranspozony, což jsou úseky DNA schopné se přepsat do RNA a pak zpětně přepsat do DNA; jejich činností vzniklo asi 42 % lidského genomu.[61]
Geny kódující proteiny často obsahují introny, nekódující oblasti, které musí být v průběhu zrání mRNA vystřihnuty. V některých případech je vystřihnutí katalyzováno samotným intronem, jedná se tedy o sebevystřihující introny. Ty velmi často kódují proteiny, které jim umožňuje šířit se genomem hostitele. Podle mechanismu sestřihu se rozlišují na typ I, který následně pro své množení zneužívá mechanismus opravy DNA tak, že „opraví“ úsek hostitelské DNA podle své sekvence; typ II se většinou šíří zpětným přepisem RNA→DNA.[64] Díky tomu, že jsou schopny se z mRNA vystřihnout, tak většinou významně nepoškozují hostitele. Je zajímavé, že mechanismus vyštěpování sebevystřihujících intronů typu II je velmi podobný eukaryotnímu spliceozomu a je pravděpodobné, že eukaryotní buňky získaly své introny tak, že se sebevystřihující introny typu II rozšířily z předchůdce mitochondrie do buňky eukaryot.[65]
Osudy RNA v buňce
Syntéza RNA a reverzní transkripce

RNA je syntetizovaná enzymy RNA polymerázami (RNApol, RNAP) podle matrice DNA, případně RNA (například u virů). RNA polymerázy s pomocí transkripčních faktorů rozpoznávají specifické úseky DNA (tzv. promotory), které označují počátek přepisu DNA na RNA. Zatímco v případě prokaryot a semiautonomních organel eukaryot jsou veškeré RNA syntetizovány jedinou RNA polymerázou, u eukaryot se nachází více typů: RNA polymeráza I syntetizující rRNA, RNA polymeráza II syntetizující prekurzor mRNA, většinu snRNA a miRNA a RNA polymeráza III syntetizující tRNA a 5S rRNA. Kromě těchto tří polymeráz společných pro eukaryota je ještě známá RNA polymeráza IV a V, obě syntetizují siRNA v rostlinných buňkách. RNA může být syntetizována i podle RNA templátu pomocí RNA dependentních RNA polymeráz, které jsou schopny replikovat viry, jejichž genom je tvořen právě RNA.
Syntéza RNA pomáhá i v procesu replikace DNA – DNA polymeráza není schopna započít syntézu nového vlákna, proto speciální DNA dependentní RNA polymeráza zvaná primáza vytvoří krátký úsek RNA komplementární s DNA (takzvaný primer), na který může DNA polymeráza navázat. RNA je následně z DNA odstraněna a nahrazena DNA.
RNA hraje také roli v případě syntézy DNA v procesu nazývaném reverzní transkripce, ve kterém slouží jako templát. Eukaryotní buňky využívají reverzní transkripci pro vytvoření svých telomer, což repetitivní oblasti chránící koncové oblasti chromozomů. Reverzní transkripci využívají také retroviry, které přepisují svůj genom kódovaný v RNA do DNA, která slouží pro integraci do hostitelského genomu; virový genom je následně vytvořen procesem transkripce.
Úpravy RNA a její lokalizace
Zatímco 5' konec mRNA u archeí není upraven[66] a u bakterií jej chrání pouze trifosfátová skupina[67] a u obou skupin je polyadenylace 3' konce signál k degradaci,[68] v případě eukaryotních mRNA a dalších molekul RNA syntetizovaných RNA polymerázou II se vyskytuje řada posttranskripčních úprav, které většinou probíhají už v průběhu syntézy, jedná se vlastně o úpravy ko-transkripční. Hlavní úpravy jsou přidání 5' čepičky, polyadenylace a splicing. Spřažení transkripce a úprav RNA zajišťuje fosforylace C-terminální domény RNA polymerázy II, jejíž stav se liší během neaktivního stavu, začátku transkripce (signál pro připojení čepičky), v průběhu elongační fáze transkripce (vyhledávání míst sestřihu) a konce transkripce (rozštěpení rostoucí mRNA a signál pro polyadenylaci). mRNA, které chybí některá z potřebných modifikací, nebo která stále obsahuje nevyštěpené introny, je degradována jadernými RNázami. Některé typy RNA (např. tRNA nebo rRNA) u všech domén života prochází složitým procesem „zrání“, při kterém dochází k jejich značným úpravám.
Protože prokaryota nemají jádro, jejich RNA je hned po syntéze dostupná v cytoplasmě a v případě mRNA překládaná. U eukaryot je RNA po kontrole své kvality exportována na cílové místo, kde vykonává svou funkci, za samotný export jsou zodpovědné proteiny, které tvoří ribonukleoproteinové komplexy s přenášenou mRNA.[69] Cílové místo určují RNA vazebné proteiny, které rozpoznávají signál na RNA směřující ji na dané místo v buňce, transportovány jsou tyto pomocné proteiny a RNA se přesunuje navázaná na ně.[70] Tento jev je nejlépe pozorovatelný u velkých a složitých buněk, jako jsou vajíčka, raná embrya nebo neurony, ale je společný pro všechny typy buněk.[71]
Vazba na proteiny

Prakticky žádná RNA se v buňce nenachází volně, RNA se většinou vyskytuje v komplexu s proteiny, tedy jako ribonukleoprotein. Proteiny vázající se na RNA mají řadu funkcí, ať už degradaci RNA nebo naopak ochranu před ní, modifikují RNA, slouží jako adaptér pro jaderný export nebo naopak udržení v jádře, případně pro skládání do složitějších ribonukleoproteinových komplexů.[72]
Proteinů vázajících RNA je celá řada, RNA-recognition motif (motiv rozeznávající RNA) je dokonce jeden z nejčastějších strukturních motivů v lidských proteinech.[73] Kromě toho, že ribonukleoproteiny jsou často velmi složité samy o sobě (například ribozom), tak se u eukaryotních organismů tyto komplexy dále shlukují do vyšších útvarů (jako je například jadérko). Předpokládá se, že vznik těchto složitějších struktur souvisí s nezbytností fyzicky oddělit a tím zefektivnit různé probíhající pochody u složitějších eukaryotních buněk[74] (podobně, jako je tomu u systému vnitřních váčků, které jsou ale odděleny membránami).
Uskladnění a kontrola mRNA v RNA granulích
mRNA, která není buňkou překládána, je často uložena v podobě RNA granulí. Ty se v cytoplazmě eukaryotních buněk často vyskytují jako dobře rozlišitelná granula tvořená RNA a proteiny. RNA granule regulují translaci, stabilitu a určují lokalizaci mRNA, která je v nich obsažená. Kromě mRNA typicky obsahují ribozomální podjednotky, translační faktory, enzymy zodpovědné za degradaci RNA, komponenty RNA interference, helikázy, strukturní proteiny a další RNA-vazebné proteiny.[75] Mezi nejdůležitější patří:
Granule v zárodečných buňkách (germ cell granules), ve kterých se vyskytují především v průběhu jejich vývoje a následně zůstávají v oocytech. Nesou vybrané molekuly mRNA kódující proteiny nezbytné pro vývoj embrya, které jsou uloženy v neaktivním stavu do doby, než budou potřeba. Kromě toho také nesou aparát RNA interference, především z rodiny piRNA, které inaktivují transpozony a brání tak genom před poškozením.[75][76]
Stresová tělíska vznikají při vystavení buněk stresu a soustřeďují v sobě mRNA kódující běžné buněčné proteiny. Nesou ribozomy bezprostředně připravené překládat nesenou mRNA, které jsou ale po dobu trvání stresu uskladněné v neaktivní stavu tak, aby nedošlo k jejich poškození. Často fyzicky interagují s P-tělísky a předávají jim RNA určenou k degradaci.[77]
P-tělíska (processing bodies) se vyskytují v somatických buňkách, kde se účastní degradace mRNA a zprostředkovávají RNA silencing pomocí miRNA. Díky schopnosti degradovat RNA slouží také ke kontrole kvality buněčných mRNA.[75]
Neuronální granule slouží neuronům pro transport mRNA k jejich axonům. Nesou ribozomy a faktory pro zahájení translace, té je ale zabráněno, dokud tyto granule nejsou dopraveny na správné místo. Neuronální granule hrají významnou roli v regeneraci neuronů a poruchy v transportu mRNA jsou spojeny s neuronálními poruchami.[78]
Degradace RNA
Degradace RNA pomocí enzymů ribonukleáz (RNáz) slouží buňce jako regulační a kontrolní mechanismus pro odstranění RNA, která je poškozená, nebo nadále není potřeba, ale také pro maturaci RNA a jako obrana proti RNA virům. Je také základem složitějších obranných strategií, jako je RNA interference. Obecně proces degradace probíhá buď od konců RNA pomocí exonukleáz (štěpící od 5' nebo 3' konce RNA), nebo pomocí endonukleáz štěpících uvnitř vlákna. V buňkách existuje celá řada RNáz s překrývajícími se aktivitami a každá RNA, která není chráněna proti jejich účinkům, je degradována.
V případě bakterií navazuje transkripce DNA bezprostředně na translaci a poločas života mRNA je mezi sekundou až hodinou. 5' konec RNA je u bakterií chráněn trifosfátovou skupinou na prvním nukleotidu a signálem pro degradaci je odštěpení dvou fosfátových zbytků a polyadenylace RNA[79] V buňkách eukaryot je poločas života mRNA delší, a to minuty až dny. Stabilita je zajištěna jednak úpravou konců – přidáním 5' čepičky a 3' polyadenylací, jednak vazbou ribonukleoproteinů bránících přístupu RNáz.
Hlavním komplexem zajišťujícím degradaci je eukaryotní exozom a u bakterií podobný, ale jednodušší, degradozom.[80] Obsahují typicky exo- i endonukleázy a polynukleotid fosforylázu štěpící krátké fragmenty RNA, vzniklé činností endonukleáz, na nukleotidy. Degradaci napomáhají helikázy zodpovědné za rozplétání sekundárních struktur na RNA. Bakteriální degradozom navíc obsahuje poly(A) polymerázu a eukaryotické exozomy proteiny zodpovědné za rozpoznání destabilizujících oblastí v RNA, například ARE elementu.
Metody práce s RNA


Práce s RNA je v mnoha ohledech podobná práci s DNA, pro izolaci RNA je tedy potřeba rozbití buněk, rozrušení membrány a denaturace proteinů pomocí detergentů. Na rozdíl od izolace DNA, při které je možné jednoduše inaktivovat enzymy štěpící DNA pomocí pouhého vyvázání dvouvazebných iontů, enzymy štěpící RNA musí drženy neaktivní pomocí chlazení nebo velmi účinných denaturačních činidel, jako je guanidium chlorid. V současnosti nejpoužívanější metody izolace RNA využívají tzv. fenol-chloroformovou extrakci právě za přítomnosti guanidium chloridu.[81] RNázy tvoří obecně velký problém při práci s RNA, protože jsou schopny vydržet vaření i autoklávování díky své schopnosti zpětného složení (renaturace).
Po izolaci celkové buněčné RNA až 98 % vzorku představuje ribozomální RNA, což komplikuje analýzu méně zastoupených typů RNA. Bohatě se vyskytující rRNA je možno enzymaticky degradovat nebo využít další techniky pro její odstranění.[36] Nejvýznamnější metodou nabohacení vybraného podtypu RNA je izolace eukaryotní mRNA pomocí oligo(dT) kotvy, která hybridizuje s poly(A) koncem mRNA, což umožňuje snadno izolovat veškeré molekuly mRNA, ale i další typy RNA, které obsahují poly(A) konec (například dlouhé nekódující RNA). Alternativní způsoby izolace žádané RNA ze směsi představuje ultracentrifugace umožňující dělit RNA nebo ribonukleoproteiny podle jejich vznášivé hustoty (podle ní jsou pojmenovány například molekuly rRNA), afinitní chromatografie, případně imunoprecipitace proteinové složky ribonukleoproteinu a následná izolace RNA.
Pro analýzu získané RNA se využívá elektroforéza, nejčastěji agarózová nebo polyakrylamidová v přítomnosti denaturačních činidel, které rozrušují sekundární strukturu RNA. Po provedení elektroforézy je možné detekovat konkrétní molekuly RNA s pomocí Northernova blotu, při kterém se z elektroforetického gelu přenese RNA na membránu, na které je RNA následně detekována pomocí hybridizace s radioaktivně nebo fluorescenčně značenou nukleovou kyselinou, která slouží jako tzv. hybridizační sonda.
Určování RNA ovšem v současnosti nejčastěji probíhá s využitím přepisu RNA do DNA pomocí reverzní transkripce, přičemž vzniká tzv. cDNA (DNA komplementární ke studované RNA). Důvodů pro tento krok je celá řada, například vyšší stabilita DNA a dále skutečnost, že enzymy pracující s DNA jsou efektivnější a méně chybují. Získanou cDNA je možné analyzovat mnoha způsoby, například určit množství původních kopií RNA ve vzorku pomocí kvantitativní PCR, což může například říct, kolik molekul mRNA pro daný gen se v buňce nachází, tedy – jak silně je gen přepisován. Sbírku všech získaných cDNA (tzv. knihovna), která představuje soubor všech genů přepisovaných do RNA (transkriptom), je možné analyzovat několika způsoby. Významný způsob analýzy velkého množství genů je DNA čip využívající nejčastěji několik tisíc různých hybridizačních sond odvozených od známých genů, při této metodě jsou molekuly cDNA ve vzorku zachyceny pomocí hybridizace k sondě umístěné na daném místě destičky („čipu“), a protože je vzorek označen (nejčastěji fluorescenčně), po odmytí zůstává signál pouze v místě, kde se cDNA navázala na sondu. Identifikaci nových genů umožňuje například analýza krátkých úseků cDNA, tzv. EST (expressed sequence tag), které jsou přečteny sekvenací. V současné době se rozšiřuje sekvenace veškeré RNA (opět ale přepsané do cDNA) přítomné buňce s použitím sekvenování nové generace, tzv. RNA-Seq (RNA Sequencing).
Dalších metod práce s RNA je nepřeberná řada, zajímavá je například fluorescenční hybridizace in situ umožňující vizualizovat pozici dané RNA v buňce, ribozomální profily umožňující zjistit, které mRNA jsou buňkou právě přepisované, nebo metody RNA interference umožňující regulovat genovou expresi v buňkách.
Odkazy
Poznámky
- ↑ Modifikace RNA se většinou z chemického pohledu popisují na úrovni ribonukleosidů, modifikace ovšem v buňce vznikají až na hotové RNA, tedy na úrovni ribonukleotidů
Reference
- ↑ a b c ZADRAŽIL, Stanislav. Ribonukleové kyseliny. Věčné „druhé“ mezi nukleovými kyselinami. Živa. 2007, čís. 3, s. 98. Dostupné online [cit. 2013-08-14].
- ↑ a b c d MAYR, Ernst. The Growth of Biological Thought: Diversity, Evolution, and Inheritance. [s.l.]: Belknap Press, 1985. 974 s. ISBN 0674364465. (anglicky)
- ↑ LEVENE, Phoebus. The Structure of Yeast Nucleic Acid: IV. Ammonia Hydrolysis. J. Biol. Chem. 1919, roč. 40, čís. 2, s. 415–424. Dostupné online [cit. 2014-03-14]. (anglicky)
- ↑ CASPERSSON, T., SCHULTZ, JACK. Pentose Nucleotides in the Cytoplasm of Growing Tissues. Nature. 1939-04-08, roč. 143, čís. 3623, s. 602–603. Dostupné online [cit. 2014-03-17]. doi:10.1038/143602c0.
- ↑ PALADE, GE. A small particulate component of the cytoplasm.. J Biophys Biochem Cytol. 1955, s. 59-68. PMID 14381428.
- ↑ CRICK, F.H.C. Ideas on Protein Synthesis [online]. Symp. Soc. Exp. Biol., 1958 [cit. 2014-04-01]. Dostupné online. (anglicky)
- ↑ The discovery of messenger RNA (mRNA) by Sydney Brenner (1927-), Francis Crick (1916-), Francois Jacob (1920-) and Jacques Monod (1910-1976). [online]. Genome News Network [cit. 2014-03-25]. Dostupné online. (anglicky)
- ↑ HOAGLAND, MB.; STEPHENSON, ML.; SCOTT, JF.; HECHT, LI.; ZAMECNIK, PC. A soluble ribonucleic acid intermediate in protein synthesis.. J Biol Chem. 1958, s. 241-57. PMID 13538965.
- ↑ a b NIRENBERG, MW.; MATTHAEI, JH. The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides.. Proc Natl Acad Sci U S A. Oct 1961, roč. 47, s. 1588–602. PMID 14479932.
- ↑ OCHOA, Severo. Enzymatic synthesis of ribonucleic acid Nobel Lecture [online]. Nobelprize.org [cit. 2014-03-28]. Dostupné online. (anglicky)
- ↑ HURWITZ, J. The discovery of RNA polymerase.. J Biol Chem. Dec 2005, roč. 280, čís. 52, s. 42 477 – 42485. doi:10.1074/jbc.X500006200. PMID 16230341.
- ↑ FREDHOLM, Lotta. Crack the Code - How the Code was Cracked [online]. Nobelprize.org [cit. 2014-03-29]. Dostupné online. (anglicky)
- ↑ a b RAJBHANDARY, UL.; KÖHRER, C. Early days of tRNA research: discovery, function, purification and sequence analysis.. J Biosci. Oct 2006, roč. 31, čís. 4, s. 439–51. PMID 17206064.
- ↑ CLARK, BF. The crystal structure of tRNA.. J Biosci. Oct 2006, roč. 31, čís. 4, s. 453–7. PMID 17206065.
- ↑ a b HOLLEY, RW.; APGAR, J.; EVERETT, GA., et al. STRUCTURE OF A RIBONUCLEIC ACID.. Science. Mar 1965, roč. 147, čís. 3664, s. 1462–5. PMID 14263761.
- ↑ FIERS, W.; CONTRERAS, R.; DUERINCK, F., et al. Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene.. Nature. Apr 1976, roč. 260, čís. 5551, s. 500–7. PMID 1264203.
- ↑ SZATHMÁRY, E. The origin of the genetic code: amino acids as cofactors in an RNA world.. Trends Genet. Jun 1999, roč. 15, čís. 6, s. 223–9. PMID 10354582.
- ↑ TEMIN, HM.; MIZUTANI, S. RNA-dependent DNA polymerase in virions of Rous sarcoma virus.. Nature. Jun 1970, roč. 226, čís. 5252, s. 1211–3. PMID 4316301.
- ↑ a b c d CECH, TR. Structural biology. The ribosome is a ribozyme.. Science. Aug 2000, roč. 289, čís. 5481, s. 878–9. PMID 10960319.
- ↑ a b KIKOVSKA, E.; SVÄRD, SG.; KIRSEBOM, LA. Eukaryotic RNase P RNA mediates cleavage in the absence of protein.. Proc Natl Acad Sci U S A. Feb 2007, roč. 104, čís. 7, s. 2062–7. doi:10.1073/pnas.0607326104. PMID 17284611.
- ↑ a b BURNETT, JC.; ROSSI, JJ. RNA-based therapeutics: current progress and future prospects.. Chem Biol. Jan 2012, roč. 19, čís. 1, s. 60–71. doi:10.1016/j.chembiol.2011.12.008. PMID 22284355.
- ↑ Macugen pegaptanib [online]. European Medicines Agency [cit. 2014-04-01]. Dostupné v archivu pořízeném dne 2013-12-13. (anglicky)
- ↑ KEEFE, AD.; PAI, S.; ELLINGTON, A. Aptamers as therapeutics.. Nat Rev Drug Discov. Jul 2010, roč. 9, čís. 7, s. 537–50. doi:10.1038/nrd3141. PMID 20592747.
- ↑ Vitravene fomivirsen) [online]. drugs.com [cit. 2014-04-01]. Dostupné online. (anglicky)
- ↑ HAIR, P.; CAMERON, F.; MCKEAGE, K. Mipomersen sodium: first global approval.. Drugs. Apr 2013, roč. 73, čís. 5, s. 487–93. doi:10.1007/s40265-013-0042-2. PMID 23564617.
- ↑ PEREIRA, TC.; LOPES-CENDES, I. Emerging RNA-based drugs: siRNAs, microRNAs and derivates.. Cent Nerv Syst Agents Med Chem. Sep 2012, roč. 12, čís. 3, s. 217–32. PMID 22697266.
- ↑ MIKKOLA, Satu, Stenman, Eeva; Nurmi, Kirsi; Yousefi-Salakdeh, Esmail; Strömberg, Roger; Lönnberg, Harri. The mechanism of the metal ion promoted cleavage of RNA phosphodiester bonds involves a general acid catalysis by the metal aquo ion on the departure of the leaving group. Journal of the Chemical Society, Perkin Transactions 2. 1999-01-01, čís. 8, s. 1619–1626. doi:10.1039/a903691a.
- ↑ KISS, T. NEW EMBO MEMBER'S REVIEW: Small nucleolar RNA-guided post-transcriptional modification of cellular RNAs. The EMBO Journal. 2001, roč. 20, čís. 14, s. 3617–3622. doi:10.1093/emboj/20.14.3617.
- ↑ CANTARA, W. A., Crain, P. F.; Rozenski, J.; McCloskey, J. A.; Harris, K. A.; Zhang, X.; Vendeix, F. A. P.; Fabris, D.; Agris, P. F. The RNA modification database, RNAMDB: 2011 update. Nucleic Acids Research. 2010-11-10, roč. 39, čís. Database, s. D195–D201. doi:10.1093/nar/gkq1028.
- ↑ ZINSHTEYN, Boris, Nishikura, Kazuko. Adenosine-to-inosine RNA editing. Wiley Interdisciplinary Reviews: Systems Biology and Medicine. 2009-09-01, roč. 1, čís. 2, s. 202–209. doi:10.1002/wsbm.10.
- ↑ SQUIRES, JE.; PATEL, HR.; NOUSCH, M., et al. Widespread occurrence of 5-methylcytosine in human coding and non-coding RNA.. Nucleic Acids Res. Jun 2012, roč. 40, čís. 11, s. 5023–33. Dostupné online. doi:10.1093/nar/gks144. PMID 22344696.
- ↑ HALL, Kathleen B. RNA in motion. Current Opinion in Chemical Biology. 2008-12-01, roč. 12, čís. 6, s. 612–618. doi:10.1016/j.cbpa.2008.09.033.
- ↑ The Nobel Prize in Chemistry 2009 [online]. http://www.nobelprize.org [cit. 2014-04-06]. Dostupné online. (anglicky)
- ↑ MATTICK, J. S. Non-coding RNAs: the architects of eukaryotic complexity. EMBO Reports. 2001, roč. 2, čís. 11, s. 986–991. doi:10.1093/embo-reports/kve230.
- ↑ JACKMAN, Jane E., Alfonzo, Juan D. Transfer RNA modifications: nature's combinatorial chemistry playground. Wiley Interdisciplinary Reviews: RNA. 2013-01-01, roč. 4, čís. 1, s. 35–48. doi:10.1002/wrna.1144.
- ↑ a b PEANO, Clelia, Pietrelli, Alessandro; Consolandi, Clarissa; Rossi, Elio; Petiti, Luca; Tagliabue, Letizia; De Bellis, Gianluca; Landini, Paolo. An efficient rRNA removal method for RNA sequencing in GC-rich bacteria. Microbial Informatics and Experimentation. 2013-01-01, roč. 3, čís. 1, s. 1. doi:10.1186/2042-5783-3-1.
- ↑ GUENEAU DE NOVOA, P.; WILLIAMS, KP. The tmRNA website: reductive evolution of tmRNA in plastids and other endosymbionts.. Nucleic Acids Res. Jan 2004, roč. 32, čís. Database issue, s. D104-8. doi:10.1093/nar/gkh102. PMID 14681369.
- ↑ KEILER, KC. Biology of trans-translation.. Annu Rev Microbiol. 2008, roč. 62, s. 133–51. Dostupné online. doi:10.1146/annurev.micro.62.081307.162948. PMID 18557701.
- ↑ NAGAI, K. NEW EMBO MEMBER'S REVIEW: Structure, function and evolution of the signal recognition particle. The EMBO Journal. 2003-07-15, roč. 22, čís. 14, s. 3479–3485. doi:10.1093/emboj/cdg337.
- ↑ FRIEDMAN, R. C., Farh, K. K.-H.; Burge, C. B.; Bartel, D. P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Research. 2008-10-29, roč. 19, čís. 1, s. 92–105. doi:10.1101/gr.082701.108.
- ↑ LI, Chunsheng, Feng, Yi; Coukos, George; Zhang, Lin. Therapeutic MicroRNA Strategies in Human Cancer. The AAPS Journal. 2009-10-29, roč. 11, čís. 4, s. 747–757. doi:10.1208/s12248-009-9145-9.
- ↑ PADDISON, P. J. Short hairpin RNAs (shRNAs) induce sequence-specific silencing in mammalian cells. Genes & Development. 2002, roč. 16, čís. 8, s. 948–958. doi:10.1101/gad.981002.
- ↑ HORVATH, P.; BARRANGOU, R. CRISPR/Cas, the immune system of bacteria and archaea.. Science. Jan 2010, roč. 327, čís. 5962, s. 167–70. doi:10.1126/science.1179555. PMID 20056882.
- ↑ TOOR, N., Keating, K. S.; Fedorova, O.; Rajashankar, K.; Wang, J.; Pyle, A. M. Tertiary architecture of the Oceanobacillus iheyensis group II intron. RNA. 2009-12-01, roč. 16, čís. 1, s. 57–69. doi:10.1261/rna.1844010.
- ↑ ZHANG, B.; CECH, TR. Peptide bond formation by in vitro selected ribozymes.. Nature. Nov 1997, roč. 390, čís. 6655, s. 96–100. doi:10.1038/36375. PMID 9363898.
- ↑ WOCHNER, A., Attwater, J.; Coulson, A.; Holliger, P. Ribozyme-Catalyzed Transcription of an Active Ribozyme. Science. 2011-04-07, roč. 332, čís. 6026, s. 209–212. doi:10.1126/science.1200752.
- ↑ CHEN, Z.; LIU, N.; ZHU, G., et al. Targeting of the anti-apoptotic gene survivin in human thyroid carcinoma.. Int J Mol Med. Sep 2012, roč. 30, čís. 3, s. 465–72. Dostupné online. doi:10.3892/ijmm.2012.1046. PMID 22751750.
- ↑ KHAN, AU. Ribozyme: a clinical tool.. Clin Chim Acta. May 2006, roč. 367, čís. 1–2, s. 20–7. doi:10.1016/j.cca.2005.11.023. PMID 16426595.
- ↑ VALADKHAN, S. Role of the snRNAs in spliceosomal active site.. RNA Biol. Roč. 7, čís. 3, s. 345–53. PMID 20458185.
- ↑ TRAHAN, C., Dragon, F. Dyskeratosis congenita mutations in the H/ACA domain of human telomerase RNA affect its assembly into a pre-RNP. RNA. 2009-02-01, roč. 15, čís. 2, s. 235–243. doi:10.1261/rna.1354009.
- ↑ SERGANOV, Alexander, Huang, Lili; Patel, Dinshaw J. Structural insights into amino acid binding and gene control by a lysine riboswitch. Nature. 2008, roč. 455, čís. 7217, s. 1263–1267. doi:10.1038/nature07326.
- ↑ COCHRANE, JC.; STROBEL, SA. Riboswitch effectors as protein enzyme cofactors.. RNA. Jun 2008, roč. 14, čís. 6, s. 993–1002. doi:10.1261/rna.908408. PMID 18430893.
- ↑ BERNHARDT, HS. The RNA world hypothesis: the worst theory of the early evolution of life (except for all the others)(a).. Biol Direct. 2012, roč. 7, s. 23. Dostupné online. doi:10.1186/1745-6150-7-23. PMID 22793875.
- ↑ PERKEL, JM. Visiting noncodarnia.. Biotechniques. Jun 2013, roč. 54, čís. 6, s. 301, 303–4. doi:10.2144/000114037. PMID 23750541.
- ↑ JOHNSSON, P.; LIPOVICH, L.; GRANDÉR, D., et al. Evolutionary conservation of long non-coding RNAs; sequence, structure, function.. Biochim Biophys Acta. Mar 2014, roč. 1840, čís. 3, s. 1063–71. doi:10.1016/j.bbagen.2013.10.035. PMID 24184936.
- ↑ DERRIEN, T.; GUIGÓ, R.; JOHNSON, R. The Long Non-Coding RNAs: A New (P)layer in the Dark Matter.. Front Genet. 2011, roč. 2, s. 107. doi:10.3389/fgene.2011.00107. PMID 22303401.
- ↑ DERRIEN, T.; JOHNSON, R.; BUSSOTTI, G., et al. The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression.. Genome Res. Sep 2012, roč. 22, čís. 9, s. 1775–89. doi:10.1101/gr.132159.111. PMID 22955988.
- ↑ a b MAO, YS.; ZHANG, B.; SPECTOR, DL. Biogenesis and function of nuclear bodies.. Trends Genet. Aug 2011, roč. 27, čís. 8, s. 295–306. doi:10.1016/j.tig.2011.05.006. PMID 21680045.
- ↑ a b c d IP, JY.; NAKAGAWA, S. Long non-coding RNAs in nuclear bodies.. Dev Growth Differ. Jan 2012, roč. 54, čís. 1, s. 44–54. doi:10.1111/j.1440-169X.2011.01303.x. PMID 22070123.
- ↑ NAKAGAWA, S.; KAGEYAMA, Y. Nuclear lncRNAs as epigenetic regulators-beyond skepticism.. Biochim Biophys Acta. Mar 2014, roč. 1839, čís. 3, s. 215–22. doi:10.1016/j.bbagrm.2013.10.009. PMID 24200874.
- ↑ a b c INTERNATIONAL HUMAN GENOME SEQUENCING CONSORTIUM. Initial sequencing and analysis of the human genome. Nature. 2001-02-15, roč. 409, čís. 6822, s. 860–921. doi:10.1038/35057062.
- ↑ BELSHAW, R.; PEREIRA, V.; KATZOURAKIS, A., et al. Long-term reinfection of the human genome by endogenous retroviruses.. Proc Natl Acad Sci U S A. Apr 2004, roč. 101, čís. 14, s. 4894–9. doi:10.1073/pnas.0307800101. PMID 15044706.
- ↑ DUPRESSOIR, A.; LAVIALLE, C.; HEIDMANN, T. From ancestral infectious retroviruses to bona fide cellular genes: role of the captured syncytins in placentation.. Placenta. Sep 2012, roč. 33, čís. 9, s. 663–71. doi:10.1016/j.placenta.2012.05.005. PMID 22695103.
- ↑ EDGELL, DR.; CHALAMCHARLA, VR.; BELFORT, M. Learning to live together: mutualism between self-splicing introns and their hosts.. BMC Biol. 2011, roč. 9, s. 22. doi:10.1186/1741-7007-9-22. PMID 21481283.
- ↑ ROGOZIN, IB.; CARMEL, L.; CSUROS, M., et al. Origin and evolution of spliceosomal introns.. Biol Direct. 2012, roč. 7, s. 11. doi:10.1186/1745-6150-7-11. PMID 22507701.
- ↑ BROWN, JW.; REEVE, JN. Polyadenylated, noncapped RNA from the archaebacterium Methanococcus vannielii.. J Bacteriol. Jun 1985, roč. 162, čís. 3, s. 909–17. Dostupné online. PMID 2581934.
- ↑ DEANA, A.; CELESNIK, H.; BELASCO, JG. The bacterial enzyme RppH triggers messenger RNA degradation by 5' pyrophosphate removal.. Nature. Jan 2008, roč. 451, čís. 7176, s. 355–8. doi:10.1038/nature06475. PMID 18202662.
- ↑ CAVICCHIOLI, Ricardo. Archaea : molecular and cellular biolog. Washington, DC: ASM Press, 2007. ISBN 978-1-55581-391-8. Kapitola RNA processing.
- ↑ HOCINE, S.; SINGER, RH.; GRÜNWALD, D. RNA processing and export.. Cold Spring Harb Perspect Biol. Dec 2010, roč. 2, čís. 12, s. a000752. doi:10.1101/cshperspect.a000752. PMID 20961978.
- ↑ MARTIN, KC.; EPHRUSSI, A. mRNA localization: gene expression in the spatial dimension.. Cell. Feb 2009, roč. 136, čís. 4, s. 719–30. doi:10.1016/j.cell.2009.01.044. PMID 19239891.
- ↑ BLOWER, MD. Molecular insights into intracellular RNA localization.. Int Rev Cell Mol Biol. 2013, roč. 302, s. 1–39. doi:10.1016/B978-0-12-407699-0.00001-7. PMID 23351709.
- ↑ ROUGEMAILLE, M.; VILLA, T.; GUDIPATI, RK., et al. mRNA journey to the cytoplasm: attire required.. Biol Cell. Jun 2008, roč. 100, čís. 6, s. 327–42. doi:10.1042/BC20070143. PMID 18479253.
- ↑ CLÉRY, A.; BLATTER, M.; ALLAIN, FH. RNA recognition motifs: boring? Not quite.. Curr Opin Struct Biol. Jun 2008, roč. 18, čís. 3, s. 290–8. doi:10.1016/j.sbi.2008.04.002. PMID 18515081.
- ↑ COLLINS, LJ.; KURLAND, CG.; BIGGS, P., et al. The modern RNP world of eukaryotes.. J Hered. Roč. 100, čís. 5, s. 597–604. doi:10.1093/jhered/esp064. PMID 19643816.
- ↑ a b c ANDERSON, P.; KEDERSHA, N. RNA granules.. J Cell Biol. Mar 2006, roč. 172, čís. 6, s. 803–8. Dostupné online. doi:10.1083/jcb.200512082. PMID 16520386.
- ↑ VORONINA, E.; SEYDOUX, G.; SASSONE-CORSI, P., et al. RNA granules in germ cells.. Cold Spring Harb Perspect Biol. Dec 2011, roč. 3, čís. 12. doi:10.1101/cshperspect.a002774. PMID 21768607.
- ↑ KEDERSHA, N.; STOECKLIN, G.; AYODELE, M., et al. Stress granules and processing bodies are dynamically linked sites of mRNP remodeling.. J Cell Biol. Jun 2005, roč. 169, čís. 6, s. 871–84. Dostupné online. doi:10.1083/jcb.200502088. PMID 15967811.
- ↑ WANG, W.; VAN NIEKERK, E.; WILLIS, DE., et al. RNA transport and localized protein synthesis in neurological disorders and neural repair.. Dev Neurobiol. Aug 2007, roč. 67, čís. 9, s. 1166–82. Dostupné online. doi:10.1002/dneu.20511. PMID 17514714.
- ↑ KABERDIN, VR.; SINGH, D.; LIN-CHAO, S. Composition and conservation of the mRNA-degrading machinery in bacteria.. J Biomed Sci. 2011, roč. 18, s. 23. doi:10.1186/1423-0127-18-23. PMID 21418661.
- ↑ CARPOUSIS, AJ. The RNA degradosome of Escherichia coli: an mRNA-degrading machine assembled on RNase E.. Annu Rev Microbiol. 2007, roč. 61, s. 71–87. Dostupné online. doi:10.1146/annurev.micro.61.080706.093440. PMID 17447862.
- ↑ CHOMCZYNSKI, P.; SACCHI, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on.. Nat Protoc. 2006, roč. 1, čís. 2, s. 581–5. doi:10.1038/nprot.2006.83. PMID 17406285.
Literatura
- ZADRAŽIL, Stanislav. Ribonukleové kyseliny. Věčné „druhé“ mezi nukleovými kyselinami. Živa. 2007, čís. 3, s. 98. Dostupné online [cit. 2013-08-14].
- HARTMANN, Roland K. (Roland Karl). Handbook of RNA biochemistry. Weinheim: Wiley-VCH, 2005. Dostupné online. ISBN 978-3-527-30826-2.
- ELLIOTT, David; LADOMERY, Michael. Molecular biology of RNA. Oxford ; New York: Oxford University Press, 2011. ISBN 978-0-19-928837-3.
Související články
- Nukleová kyselina
- Molekulární biologie
- Centrální dogma molekulární biologie
- Genetický kód
- RNA vazebný protein
- Ribonukleoprotein
Externí odkazy
 Obrázky, zvuky či videa k tématu RNA na Wikimedia Commons
Obrázky, zvuky či videa k tématu RNA na Wikimedia Commons  Slovníkové heslo RNA ve Wikislovníku
Slovníkové heslo RNA ve Wikislovníku- http://openwetware.org/wiki/RNA
Média použitá na této stránce
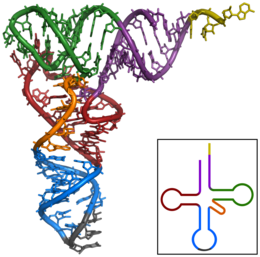
Autor: Yikrazuul, Licence: CC BY-SA 3.0
X-ray structure of the tRNAPhe from yeast. Data was obtained by PDB: 1ehz and rendered with PyMOL.
- violet: acceptor stem
- wine red: D-loop
- blue: anticodon loop
- orange: variable loop
- green: TPsiC-loop
- yellow: CCA-3' of the acceptor stem
- grey: anticodon
Curly cat
(c) User:Ppgardne, CC BY-SA 3.0
miR-137 microRNA secondary structure and sequence conservation
Autor:
- Ribosome_mRNA_translation_es.svg: LadyofHats. Translation by User: Parri.
- derivative work: Faskal
Diagram showing how the translation of the mRNA and the synthesis of proteins is made by ribosomes.
Autor: Faskal, Licence: CC BY-SA 3.0
A scheme of Marshall W. Nirenberg's experiment, which elucidated the first "letter" of the genetic code. He used poly-uracil (shown in a first row) as a template for in-vitro protein synthesis; he obtained polypeptide composed only of phenylalanine (second row).
Autor: Vossman, Licence: CC BY-SA 3.0
A hairpin loop from a pre-mRNA. Notice its bases (light green) and backbone (sky blue). NMR structure of the central region of the human GluR-B R/G pre-mRNA, from the protein data bank ID 1ysv. Taken from w:PDB ids 1ysv model 1 imaged using w:UCSF Chimera.
Autor: RNAMacGyver, Licence: CC BY-SA 3.0
Structure of a bacterial ribonuclease P holoenzyme in complex with tRNA.
Autor: B Reinius & C Shi, Licence: CC BY-SA 3.0
The figure shows confocal images from a combined RNA-DNA FISH experiment for Xist in female mouse fibroblast cells (Female-biased expression of long non-coding RNAs in domains that escape X-inactivation in mouse, B Reinius et al 2010, BMC Genomics (6), http://www.biomedcentral.com/1471-2164/11/614/abstract). The figure was adapted from Figure 5 in the original publication and contrast was adjusted to make the figure clear in thumbnail format. Open access: Unrestricted use, distribution and reproduction in any medium is permitted, provided the original article is properly cited.
Autor: Ryan Jeffs, Licence: CC BY-SA 3.0
ViewRNA assay for detection of miR-133 microRNA (green) and myogenin mRNA (red) in differentiating C2C12 cells.
Autor: Faskal, Licence: CC BY-SA 3.0
Tandem RNA-recognition motifs in human TDP-43, modelled using PyMol, PDBID:4BS2, RNA in cyan, helixes yellow, beta-sheets in slate
Já, držitel autorských práv k tomuto dílu, ho tímto zveřejňuji za podmínek následující licence:
Autor: Faskal, Licence: CC BY-SA 3.0
Dvouvláknová RNA tvořená polyuracilem a polyadeninem. Vytvořeno pomocí Pymol podle PDBID:1H1K
Autor: Fdardel, Licence: CC BY-SA 3.0
Structure of the biotin RNA aptamer (yellow) complexed with biotin - created with pymol using PBD entry 1F27
(c) Narayanese, CC BY-SA 3.0
Diagram to illustrate 5' to 3' directionality in a nucleic acid (shown here in an RNA structure).
Autor: Yikrazuul, Licence: CC BY-SA 3.0
large ribosomal subunit (50S) of Haloarcula marismortui, facing the 30S subunit. The ribosomal proteins are shown in blue, the rRNA in ochre, the active site (A 2486) in red. Data were taken from PDB: 3CC2, redered with PyMOL.
Autor: Dovelike, Licence: CC BY-SA 3.0
simple DNA transcription diagram featuring the sense and antisense strands of DNA and the production of messenger RNA via RNA polymerase.
Autor: Yikrazuul, Licence: CC BY-SA 3.0
structure of a pseudoknot of pdb-file 1YMO. (A) sticks (B) backbone