Tandemová hmotnostní spektrometrie

Tandemová hmotnostní spektrometrie, zkráceně MS/MS nebo MS2 je druh instrumentální analýzy, při kterém se propojuje více hmotnostních spektrometrů za účelem přidání dalšího reakčního kroku a zlepšení parametrů analýzy chemických vzorků.[1] Často se používá k analýze biomolekul, jako jsou bílkoviny a peptidy.
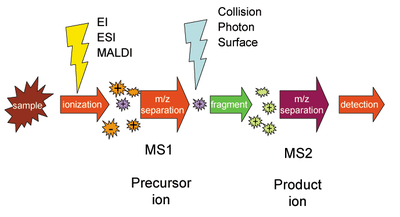
Molekuly vzorku se ionizují a první spektrometr (MS1) oddělí vzniklé ionty podle jejich poměru hmotnosti a náboje (značeného m/z nebo m/Q). Ionty o určitých hodnotách m/z-ratio jsou odděleny a následně přeměněny na fragmentované ionty, například srážkami indukovanou disociací, reakcemi iontů s molekulami nebo fotodisociací. Tyto fragmenty poté vstupují do druhého hmotnostního spektrometru (MS2), kde se opět rozdělí podle m/z a jsou detekovány. Fragmentační krok umožňuje identifikaci a oddělení iontů s velmi podobnými hodnotami m/z v běžných hmotnostních spektrometrech.
Struktura
K druhům tandemové hmotnostní spektrometrie na základě uspořádání patří například trojitý kvadrupólový hmotnostní spektrometr (qqq), čtyřnásobný spektrometr doby letu (Q-tof) a hybridní hmotnostní spektrometr.
Trojitý kvadrupólový hmotnostní spektrometr
Trojité kvadrupólové hmotnostní spektrometry používají první a třetí kvadrupól jako hmotnostní filtry. Při průchodu analytů druhým kvadrupólem dochází k fragmentaci srážkami jejich molekul s plynem. Toto uspořádání je nejběžnější ve farmaceutickém průmyslu.
Kvadrupólový spektrometr doby letu
Kvadrupólové spektrometry doby letu (Q-tof) spojují hmotnostní spektrometrii doby letu a kvadrupólové hmotnostní spektrometry, čímž se dosahuje vysoké hmotnostní přesnosti u vytvořených iontů a využitelnosti fragmentačních experimentů. Poměr (m/z) je zde určován měřením doby letu iontů.
Hybridní hmotnostní spektrometr
Hybridní hmotnostní spektrometr je soustava vzniklá propojením více než dvou hmotnostních spektrometrů.
Přístroje
Tandemová hmotnostní spektrometrie v prostoru

Při tandemové hmotnostní spektrometrii v prostoru jsou separační zařízení fyzicky oddělená, i když jsou jednotlivé prvky stále propojené, aby bylo zachováno vysoké vakuum. Těmito prvky mohou být sektory, kvadrupóly nebo spektrometry doby letu. Je-li použito více kvadrupólových spektrometrů, tak mohou sloužit jako hmotnostní analyzátory i srážkové komory.
Pro hmotnostní analyzátory se obvykle používá toto značení Q – kvadrupólový hmotnostní analyzátor; q – radiofrekvenční kolizní kvadrupól; TOF – hmotnostní analyzátor doby letu; B – magnetický sektor a E – elektrický sektor. Značení je možné při označování kombinovaných zařízení spojovat, například QqQ' – trojitý kvadrupólový hmotnostní spektrometr; QTOF – kvadrupólový hnotnostní spektrometr doby letu (také značený QqTOF) a BEBE – čtyřsektorový hmotnostní spektrometr (s obrácenou geometrií).
Tandemová hmotnostní spektrometrie v čase

U tandemové hmotnostní spektrometrie v čase separace probíhá společně se zachytáváním iontů na stejném místě, přičemž dojde k několika separačním krokům. Používají se například kvadrupólové iontové pasti a iontová cyklotronová rezonance s Fourierovou transformací (FTICR).[2] Analýza se může skládat z několika kroků, pak se někdy používají zkratky ve tvaru MSn;[3] například MS3 označuje třífázovou separaci.
Přístroje na tandemovou hmotnostní spektrometrii v čase nevyužívají níže popsané režimy, ale obvykle získávají data skenováním prekurzorů iontů v rámci celého spektra. Každé uspořádání zařízení představuje jedinečný způsob analýzy.
Tandemová MS/MS v prostoru
Při provádění tandemové MS v prostoru existuje několik režimů provozu soustavy. Existuje řada různých druhů seřazení přístrojů a každá má své využití a poskytuje jiné údaje. Tandemová hmotnostní spektrometrie v prostoru spojuje dva přístroje, které měří spektra ve stejném rozsahu, ovšem s kontrolovanou tvorbou fragmentů, zatímco tandemová MS v čase využívá iontové pasti.
Existují čtyři hlavní uspořádání MS/MS: detekce prekurzorových iontů, detekce produktových iontů, ztráty neutrálních částic a sledování vybraných reakcí.
U detekce prekurzorových iontů se ionty produktu třídí ve druhém hmotnostním analyzátoru a ionty prekurzoru v prvním.
V detekci produktových iontů se třídí ionty prekurzoru v prvním spektrometru, což dovoluje třídění fragmentů a dalších vzniklých částic ve druhém hmotnostním analyzátoru a detekci detektorem umístěným za druhým hmotnostním analyzátorem. Tato metoda se používá k identifikaci procesů využívaných k určení množství látek pomocí tandemové hmotnostní spektrometrie.
Při zkoumání ztráty neutrálních částic zkoumá první hmotnostní analyzátor všechny částice. Druhý analyzátor pak zkoumá částice s určitým posunem oproti prvnímu analyzátoru.[4] Tento posun odpovídá ztrátě neutrální částice. Při zkoumání konstantní ztráty se sledují všechny prekurzory, u kterých dochází k oddělení konkrétní částice. Oba hmotnostní analyzátory neprovádějí detekci současně, ale s určitým posunem odpovídajícím hmotnosti příslušné neutrální částice. Podobně jako u zkoumání prekurzorových iontů se tento postup dá použít k selektivní identifikaci strukturně podobných sloučenin.
Při zkoumání vybraných reakcí jsou oba analyzátory nastaveny na stejnou hmotnost. Jedná se o selektivní analytickou metodu, která vykazuje vysokou citlivost.[5]
Fragmentace
Fragmentace iontů v plynné fázi je pro tandemovou hmotnostní spektrometrii důležitá; dochází k ní v různých fázích analýzy. K fragmentaci iontů se používají různé postupy, které se mohou lišit způsoby fragmentace a tedy i druhem informací o struktuře a složení molekul, která je možné pomocí nich získat.
Fragmentace ve zdroji
Často je samotná ionizace dostatečně silná na to, aby se fragmenty vytvářely už ve hmotnostním spektrometru. Pokud ionty produktu zůstanou v nerovnovážném stavu dostatečně dlouhou dobu před autodisociací, pak se jedná o metastabilní fragmentaci.[6] Fragmentaci ve zdroji lze v tandemové hmotnostní spektrometrii použít prostřednictvím metody EISA, při níž fragmentace přímo odpovídá datům z tandemové hmotnostní spektrometrie.[7]
Fragmenty pozorované při EISA mají vyšší intenzitu signálu než běžné fragmenty, které ztrácejí energii srážkami.[8]
EISA umožňuje získat data o fragmentaci u MS1 hmotnostních analyzátorů, jako jsou přístroje založené na době letu a jednoduchá kvadrupólová zařízení. Fragmentace uvnitř zdroje se často používá k umožnění dvoukrokové fragmentace u pseudo MS3 experimentů.[9]
Srážkami indukovaná disociace
Postzdrojová fragmentace se v tandemové hmotnostní spektrometrii používá nejčastěji. Energie se dodává iontům, které jsou již většinou vibračně excitované srážkami s neutrálními atomy nebo molekulami, absorpcí záření, nebo přenosy či záchyty elektronů ionty s vícenásobnými náboji. Srážkami indukovaná disociace, také někdy nazývaná aktivovaná disociace, spočívá ve srážkách iontů s neutrálními částicemi v plynné fázi a následné disociaci iontů.[10][11]
Tento proces lze popsat následujícím schématem:
kde se ion AB+ sráží s neutrální částicí M a poté se rozpadá. S ohledem na různé konfigurace přístroje se objevují dva hlavní typy disociace: (i) paprskový (kdy jsou ionty prekurzoru fragmentovány během letu)[12] a (ii) iontová past (při které jsou ionty prekurzoru nejprve zachyceny, a až poté fragmentovány).[13][14]
Třetím, novějším, typem srážkami indukované disociace je srážková disociace při vyšších energiích (HCD). Jedná se o techniku specifickou pro orbitrapové hmotnostní spektrometry, u nichž fragmentace probíhá mimo iontovou past,[15][16] proces probíhá v HCD komorách (v některých přístrojích nazývaných "iontové směrovací multipóly").[17]
HCD je fragmentační metoda typu iontové pasti, která má určité vlastnosti odpovídající paprskovému typu.[18][19] Existují volně dostupné databáze pro tandemovou hmotnostní spektrometrii, jako například METLIN, zahrnující 850 000 molekulových standardů s příslušnými experimentálními CID MS/MS daty);[20] obvykle se využívají k identifikaci menších molekul.
Záchyt a přenos elektronů
Energie uvolněná při záchytu elektronů na vícenásobně nabitých iontech či při přenosu elektronů mezi nimi může způsobovat fragmentaci.
Disociace záchytem elektronu
Pokud je elektron zachycen vícenásobně nabitým kladným iontem, tak se uvolní Coulombova energie; příslušný proces se nazývá disociace záchytem elektronu (ECD):[21]
kde M je vícekrát protonovaná molekula.
Disociace přenosem elektronů
Navázání elektronu při reakci dvou iontů se nazývá disociace přenosem elektronu (ETD).[22][23]
Podobně jako u disociace záchytem elektronu ETD vyvolává fragmentaci kationtů (například peptidových nebo bílkovinových) přenášením elektronů na tyto částice. Tuto metodu objevili Donald F. Hunt, Joshua Coon, John E. P. Syka a Jarrod Marto na Univerzitě ve Virginii.[24]
ETD nevyužívá volné elektrony, ale radikálové anionty (odvozené například od anthracenu či azobenzenu):
kde A je anion.[25]
ETD způsobuje štěpení náhodně podél hlavního peptidového řetězce (za vzniku c- a z iontů), zatímco vedlejší řetězce a modifikace, jako je fosforylace, jí zasaženy nejsou. Technika dobře funguje pouze u iontů s většími náboji (z>2), ovšem podobně jako u srážkami vyvolávané disociace (CID) lze pomocí ETD fragmentovat i větší peptidy až bílkoviny, což má využití v proteomice. Podobně jako ECD je ETD dobře využitelnou metodou u peptidů s posttranslačními modifikacemi, jako je fosforylace.[26]
Disociace přenosem elektronů a disociace srážkami s vyšší energií (EThcD) je kombinací ETD a HCD, kde peptidový prekurzor vstupuje do iontovo/iontových reakcí s anionty fluoranthenu v lineární iontové pasti, která vytváří c- a z-ionty.[22][27]
V druhém kroku se použije HCD fragmentace na všechny ionty vytvořené pomocí ETD, čímž vzniknou b- a y ionty, jež jsou nakonec analyzovány v orbitrapovém analyzátoru.[15] Při této metodě se používá dvojí fragmentace, protože se tvoří velké množství iontů a MS/MS spektra jsou tak bohatá na data využitelná při sekvencování peptidů a hledání posttranslačních modifikací.[28]
Negativní disociace s přenosem elektronů
Fragmentace také může probíhat u deprotonovaných částic, kdy z nich elektrony přecházejí na kationtové reaktanty v procesu nazývaném negativní disociace s přenosem elektronů (NETD):[29]
Po této reakci u aniontu dojde ke vnitřnímu přesmyku a fragmentaci. NETD je iont/iontovou obdobou disociace odštěpením elektronů (EDD).
NETD je možné použít k fragmentaci peptidů podél hlavního řetězce na vazbách Cα-C. Vzniklé fragmenty jsou obvykle typu a•- a x.
Disociace odštěpením elektronů
Disociace odštěpením elektronů (EDD) slouží k fragmentaci aniontů.[30]
Jedná se o protipól disociace záchytem elektronů. Záporně nabité ionty se aktivují ozářením elektrony se středně vysokými kinetickými energiemi. Dojde tak k odštěpení elektronů z původní iontové molekuly a v důsledku rekombinace k disociaci.
Disociace přenosem náboje
Reakce kladně nabitých peptidů s kationtovými reaktanty,[31] známá jako disociace přenosem náboje (CTD),[32] byla uznána za alternativní metodu vysokoenergetické fragmentace peptidových iontů s nízkými náboji (+1 nebo +2). Navržený mechanismus CTD s využitím heliových kationtů jako reaktantů vypadá takto:
Bylo zjištěno, že CTD vyvolává Cα-C štěpení vazeb na hlavních řetězcích peptidů a vznikají při něm ionty druhu a•- a x.
Fotodisociace
Energii potřebnou k fragmentaci může dodat foton, jehož absorpcí dojde k fotodisociaci iontu podle následujícího schématu:
kde je foton absorbovaný iontem. Lze použít ultrafialové lasery, které však způsobují nadměrnou fragmentaci.[33]
Vícefotonová infračervená disociace
Fotony infračerveného záření při absorbování v dostatečném množství zahřívají ionty a způsobují jejich disociaci. Tento proces se nazývá vícefotonová infračervená disociace (IRMPD) a obvykle se provádí pomocí CO2 laseru a hmotnostního spektrometru s iontovou pastí, například FTMS.[34]
Disociace infračerveným zářením černého tělesa
Záření černého tělesa lze využít k fotodisociaci v metodě nazývané disociace infračerveným zářením černého tělesa.[35]
Při této metodě se vakuová komora hmotnostního spektrometru zahřívá a vyzařuje tak infračervené záření. Toto záření vyvolává vibrace iontů, které nakonec vedou k zániku vazeb a tvorbě fragmentů.[35][36]
Tento postup se podobá vícefotonové infračervené disociaci, při které se ovšem infračervené záření získává jiným způsobem.[11] Obvykle se používá společně s cyklotronovou rezonancí iontů s Fourierovou transformací.
Povrchem indukovaná disociace
Při povrchem indukované disociaci (SID) je fragmentace způsobována srážkami iontů s povrchem ve vysokém vakuu.[37][38]
SID lze použít k fragmentaci mnoha různých iontů. Původně se tato metoda běžně používala pouze u iontů s nízkými molekulovými hmotnostmi a s jednotkovými náboji, protože metody ionizace a hmotnostní analýzy ještě nebyly dostatečně vyvinuté ke zkoumání iontů s vysokým m/z. Postupně se však začaly používat jednovrstvé povrchy SAM tvořené CF3(CF2)10CH2CH2S na vrstvě zlata. SAM jsou pro tento účel vhodné, protože mají vysoké efektivní hmotnosti při srážkách s ionty a navíc jsou tvořeny nereaktivními fluorouhlovodíkovými řetězci, které výrazně snižují energie dopadajících iontů. Fluorouhlovodíky taktéž omezují přenos elektronů z povrchu kovu na ionty.[39]
Možností SID vytvářet stabilní subkomplexy, které poskytují využitelné informace o struktuře, nelze dosáhnout žádnou jinou známou technikou. Díky tomu, že jsou tyto komplexy stabilní a zachovávají rozdělení náboje na fragmentu, tak lze získat jedinečná spektra s komplexy majícími užší rozdělení m/z. Produkty této metody a jejich energie odpovídají vždy síle a topologii komplexu. Jedinečné vzory disociace umožňují zjistit kvaternární struktury. Symetrické rozdělení náboje a disociace jsou typické pro SID a mají za následek spektra odlišná od ostatních disociačních jednotek.[39]
SID lze využít i ve ihmotnostní spektrometrii iontové pohyblivosti (IM-MS). Objevuje se zde trojice metod, pro určení topologie, propojení podjednotek, a rozvinování struktury. Analýza rozvinování struktury je nejčastějším způsobem využití SID. U hmotnostní spektrometrie iontové pohyblivosti (IM-MS) SID slouží k disociaci zdrojů aktivovaných prekurzorů tří různých typů proteinových komplexů: C-reaktivního proteinu (CRP), transthyretinu (TTR) and konkanavalinu A (Con A). Při analýze míry rozvinování SID zobrazuje strukturu prekurzorových iontů před srážkou s povrchem. IM-MS využívá SID jako způsob potvrzení struktury každé podjednotky.[40]
Cyklotronovou rezonancí s Fourierovou transformací (FTICR) je možné získat velmi vysoké rozlišení a vysokou hmotnostní přesnost. Díky těmto vlastnostem jsou FTICR hmotnostní spektrometry užitečné v mnoha oblastech, jako jsou některé disociační experimenty,[41] jako jsou srážkami indukovaná disociace (CID) a disociace přenosem elektronů (ETD).[42] FTICR s povrchem indukovanou disociací byl využit při výzkumu fragmentace peptidů, konkrétně šlo o energetiku a kinetiku fragmentace v plynné fázi uvnitř ICR přístroje.[43]
Díky tomuto experimentu byla lépe prozkoumaná fragmentace protonovaných peptidů, peptidových iontů s lichým počtem elektronů, nekovalentních ligand-peptidových komplexů a ligandovaných shluků kovů.
Kvantitativní proteomika
Kvantitativní proteomika slouží k určení relativního nebo absolutního množství bílkovin ve vzorku.[44][45][46]
Několik kvantitativních proteomických metod je založeno na tandemové hmotnostní spektrometrii. MS/MS je důležitá při určování struktur velkých biomolekul.[47]
Jednou z těchto metod je izobarické značkování, které umožňuje současnou identifikaci a kvantifikaci proteinů z různých vzorků během jediné analýzy. Za účelem kvantifikace se peptidy značkují chemickými značkami se stejnou strukturou a molekulovou hmotností, které se však liší zapojením těžších izotopů do své struktury. Tyto značky, často označované jako tandemové hmotnostní značky, jsou vyvinuty tak, aby byly odštěpeny ve specifické oblasti pomocí srážkové disociace při vyšších energiích (HCD) v průběhu tandemové hmotnostní spektrometrie za vzniku iptů o různých hmotnostech. Kvantifikace se provádí srovnáním intenzit signálů těchto iontů v MS/MS spektru. K dostupným značkám patří iTRAQ a TMT.
Izobarické značky pro relativní a absolutní kvantifikaci (iTRAQ)

Izobarická značka pro relativní a absolutní kvantifikaci (iTRAQ) je reaktant pro tandemovou hmotnostní spektrometrii používaný k určení obsahu bílkovin z různých zdrojů při jediné analýze.[48][49][50]
V této metodě se molekuly značkují stabilními izotopy, které mohou tvořit kovalentní vazby s N-konci a aminovými skupinami v postranních řetězcích. iTRAQ reaktanty se použijí ke značkování peptidů z různých vzorků, které jsou poté spojeny a analyzovány kapalinovou chromatografií a tandemovou hmotnostní spektrometrií. Fragmentací připojené značky vznikají nízkomolekulární ionty, jež mohou být použity k relativní kvantifikaci peptidů a proteinů, ze kterých byly vytvořeny.
Tandemové hmotnostní značky (TMT)
Teandemové hmotnostní značky (TMT) je izobarická hmotnostní značka používaná při identifikaci a kvantifikaci proteinů.[51] Díky TMT reaktantům je možné současně analyzovat 2 až 11 různých vzorků peptidů získaných z buněk, tkání nebo tělních tekutin. K dispozici jsou tři druhy TMT reaktantů, přičemž každý má jinou reaktivitu: (1) reaktivní NHS estery ke značení primárních aminů (TMTduplex, TMTsixplex, TMT10plex plus TMT11-131C), (2) jodacetylové skupiny ke značení volných sulfhydrylů (iodoTMT) a (3) alkoxyaminy ke značení karbonylových skupin (aminoxyTMT).
Využití
Analýza peptidů

Tandemová hmotnostní spektrometrie může být využita při sekvenování bílkovin.[52] Zkoumané proteiny mohou být přímo aplikovány do hmotnostního analyzátoru, nebo je lze rozložit na menší peptidové řetězce a až poté analyzovat. Další možností je rozklad proteinů na peptidy ještě před separací a provedením analýzy hmotnostní spektrometrií.
Tandemová hmotnostní spektrometrie může vytvářet peptidové sekvenční značky, které lze použít k identifikaci proteinu s využitím databází.[53][54][55]
Byla vyvinuta notace k popisu fragmentů vznikajících v tandemové hmotnostní spektrometrii.[56]
Fragmenty peptidových iontů se označují a, b, nebo c, jestliže zůstává zachovaný náboj na N-konci a x, y, nebo z, když zůstává zachován náboj na C-konci. Pomocí dolního indexu se pak označí počet aminokyselinových zbytků ve fragmentu. K označení ztrát neutrálních molekul při fragmentaci vedlejších řetězců, * při odštěpení amoniaku a ° pro odštěpení vody. I když je štěpení vedlejších řetězců pro sekvenování a identifikaci peptidů nejvhodnější, tak lze při disociaci za vysokých energií pozorovat i jiné fragmentované ionty; patří sem odštěpení d, v, w a amonných iontů z postranního řetězce[57][58] i jiné sekvenčně specifické fragmentované ionty související s konkrétními aminokyselinovými zbytky.[59]
Oligosacharidy
Oligosacharidy mohou být pomocí tandemové hmotnostní spektrometrie sekvencovány podobně jako peptidy.[60]
Fragmentace většinou nastává na obou koncích glykosidové vazb (vznikají b, c, y a z ionty), ovšem za vyšších energií dochází ke štěpení i na sacharidovém kruhu (za tvorby x iontů). I zde se k označení polohy místa rozštěpení na řetězci používají dolní indexy; při popisu štěpení napříč cykly jsou používány horní indexy.[61][62]
Oligonukleotidy
Tandemová hmotnostní spektrometrie může být použita i při sekvenování DNA a RNA.[63][64]
Byla navržena notace pro fragmentaci oligonukleotidů v plynné fázi.[65]
Omezení
Tandemovou hmotnostní spektrometrii nelze použít na analýzu na úrovni jednotlivých buněk, protože na takové úrovni není dostatečně citlivá. Toto omezení je způsobeno hlavně neefektivní tvorbou iontů a jejich ztrátou působením rozpouštědel.[66]
Možnosti
Tandemová hmotnostní spektrometrie je užitečnou metodou při zkoumání proteinů, komplexů nukleoproteinů, i jiných biomolekul.[67]
Odkazy
Literatura
- McLuckey SA, Busch KL, Glish GL. Mass spectrometry/mass spectrometry: techniques and applications of tandem mass spectrometry. New York, N.Y: VCH Publishers, 1988. ISBN 978-0-89573-275-0.
- McLuckey SA, Glish GL. Mass Spectrometry/Mass Spectrometry: Techniques and Applications of Tandem. Chichester: John Wiley & Sons ISBN 978-0-471-18699-1.
- McLafferty FW. Tandem mass spectrometry. New York: Wiley, 1983. ISBN 978-0-471-86597-1.
- Sherman NE, Kinter M. Protein sequencing and identification using tandem mass spectrometry. New York: John Wiley, 2000. ISBN 978-0-471-32249-8.
Související články
Externí odkazy
 Obrázky, zvuky či videa k tématu Tandemová hmotnostní spektrometrie na Wikimedia Commons
Obrázky, zvuky či videa k tématu Tandemová hmotnostní spektrometrie na Wikimedia Commons
Reference
V tomto článku byl použit překlad textu z článku Tandem mass spectrometry na anglické Wikipedii.
- ↑ The IUPAC Compendium of Chemical Terminology: The Gold Book. Příprava vydání Victor Gold. 4. vyd. Research Triangle Park, NC: International Union of Pure and Applied Chemistry (IUPAC) Dostupné online. DOI 10.1351/goldbook.t06250. (anglicky) DOI: 10.1351/goldbook.
- ↑ R. B. Cody; B. S. Freiser. Collision-induced dissociation in a fourier-transform mass spectrometer. International Journal of Mass Spectrometry and Ion Physics. 1982, s. 199–204. DOI 10.1016/0020-7381(82)85035-3. Bibcode 1982IJMSI..41..199C.
- ↑ R. B. Cody; R. C. Burnier; C. J. Cassady; B. S. Freiser. Consecutive collision-induced dissociations in Fourier transform mass spectrometry. Analytical Chemistry. 1982-11-01, s. 2225–2228. DOI 10.1021/ac00250a021.
- ↑ John N. Louris; Larry G. Wright; R. Graham Cooks; Alan E. Schoen. New scan modes accessed with a hybrid mass spectrometer. Analytical Chemistry. 1985, s. 2918–2924. DOI 10.1021/ac00291a039.
- ↑ Edmond deHoffman; Vincent Stroobant. Mass Spectrometry: Principles and Applications. [s.l.]: Wiley, 2003. ISBN 978-0-471-48566-7. S. 133.
- ↑ The IUPAC Compendium of Chemical Terminology: The Gold Book. Příprava vydání Victor Gold. 4. vyd. Research Triangle Park, NC: International Union of Pure and Applied Chemistry (IUPAC) Dostupné online. DOI 10.1351/goldbook.p04864. (anglicky) DOI: 10.1351/goldbook.
- ↑ Xavier Domingo-Almenara; J. Rafael Montenegro-Burke; Carlos Guijas; Erica L.-W. Majumder; H. Paul Benton; Gary Siuzdak. Autonomous METLIN-Guided In-source Fragment Annotation for Untargeted Metabolomics. Analytical Chemistry. 2019-03-05, s. 3246–3253. DOI 10.1021/acs.analchem.8b03126. PMID 30681830.
- ↑ Enhanced in-Source Fragmentation Annotation Enables Novel Data Independent Acquisition and Autonomous METLIN Molecular Identification. Analytical Chemistry. 2020-04-21, s. 6051–6059. DOI 10.1021/acs.analchem.0c00409. PMID 32242660.
- ↑ R. Körner; M. Wilm; K. Morand; M. Schubert; M. Mann. Nano electrospray combined with a quadrupole ion trap for the analysis of peptides and protein digests. Journal of the American Society for Mass Spectrometry. 1996, s. 150–156. DOI 10.1016/1044-0305(95)00626-5. PMID 24203235.
- ↑ J. M. Wells; S. A. McLuckey. Methods in Enzymology. [s.l.]: [s.n.], 2005. Dostupné online. ISBN 9780121828073. DOI 10.1016/S0076-6879(05)02005-7. Kapitola Collision‐Induced Dissociation (CID) of Peptides and Proteins, s. 148–185.
- ↑ a b L. Sleno; D. A. Volmer. Ion activation methods for tandem mass spectrometry. Journal of Mass Spectrometry. 2004, s. 1091–1112. DOI 10.1002/jms.703. PMID 15481084. Bibcode 2004JMSp...39.1091S.
- ↑ Y. Xia; X. Liang; S. A. McLuckey. Ion trap versus low-energy beam-type collision-induced dissociation of protonated ubiquitin ions. Analytical Chemistry. 2006, s. 1218–1227. DOI 10.1021/ac051622b. PMID 16478115.
- ↑ Raymond E. March. An Introduction to Quadrupole Ion Trap Mass Spectrometry. Journal of Mass Spectrometry. 1997-04-01, s. 351–369. DOI 10.1002/(sici)1096-9888(199704)32:4<351::aid-jms512>3.0.co;2-y. Bibcode 1997JMSp...32..351M.
- ↑ M. Bantscheff; M. Boesche Eberhard; D. Bantscheff; T. Matthieson; G. Sweetman; B. Kuster. Robust and sensitive iTRAQ quantification on an LTQ Orbitrap mass spectrometer. Molecular & Cellular Proteomics. 2008, s. 1702–1713. DOI 10.1074/mcp.M800029-MCP200. PMID 18511480.
- ↑ a b J. V. Olsen; B. Macek; O. Lange; A. Makarov; S. Horning; M. Mann. Higher-energy C-trap dissociation for peptide modification analysis. Nature Methods. 2007, s. 709–712. DOI 10.1038/nmeth1060. PMID 17721543.
- ↑ Novel parallelized quadrupole/linear ion trap/Orbitrap tribrid mass spectrometer improving proteome coverage and peptide identification rates. Analytical Chemistry. 2013, s. 11710–11714. DOI 10.1021/ac403115c. PMID 24251866.
- ↑ N. M. Riley; M. S. Westphall; J. J. Coon. Activated Ion-Electron Transfer Dissociation Enables Comprehensive Top-Down Protein Fragmentation. Journal of Proteome Research. 2017, s. 2653–2659. DOI 10.1021/acs.jproteome.7b00249. PMID 28608681.
- ↑ N. Nagaraj; R. C. D'Souza; J. Cox; J. V. Olsen; M. Mann. Feasibility of large-scale phosphoproteomics with higher energy collisional dissociation fragmentation. Journal of Proteome Research. 2010, s. 6786–6794. DOI 10.1021/pr100637q. PMID 20873877.
- ↑ Differentiating Positional Isomers of Nucleoside Modifications by Higher-Energy Collisional Dissociation Mass Spectrometry (HCD MS). Journal of the American Society for Mass Spectrometry. 2018, s. 1745–1756. DOI 10.1007/s13361-018-1999-6. PMID 29949056. Bibcode 2018JASMS..29.1745J.
- ↑ Article Metrics - METLIN MS 2 molecular standards database: a broad chemical and biological resource [online]. Dostupné online.
- ↑ H. J. Cooper; K. Håkansson; A. G. Marshall. The role of electron capture dissociation in biomolecular analysis. Mass Spectrometry Reviews. 2005, s. 201–222. DOI 10.1002/mas.20014. PMID 15389856. Bibcode 2005MSRv...24..201C.
- ↑ a b J. E. Syka; J. J. Coon; M. J. Schroeder; J. Shabanowitz; D. F. Hunt. Peptide and protein sequence analysis by electron transfer dissociation mass spectrometry. Proceedings of the National Academy of Sciences of the United States of America. 2004, s. 9528–9533. DOI 10.1073/pnas.0402700101. PMID 15210983. Bibcode 22004PNAS..101.9528S.
- ↑ L. M. Mikesh; B. Ueberheide; A. Chi; J. J. Coon; J. E. Syka; J. Shabanowitz; D. F. Hunt. The utility of ETD mass spectrometry in proteomic analysis. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 2006, s. 1811–1822. DOI 10.1016/j.bbapap.2006.10.003. PMID 17118725.
- ↑ US7534622B2 Electron transfer dissociation for biopolymer sequence mass spectrometric analysis. worldwide.espacenet.com [online]. [cit. 2023-12-24]. Dostupné online.
- ↑ S. A. McLuckey; J. L. Stephenson. Ion/ion chemistry of high-mass multiply charged ions. Mass Spectrometry Reviews. 1998, s. 369–407. Dostupné online. DOI 10.1002/(SICI)1098-2787(1998)17:6<369::AID-MAS1>3.0.CO;2-J. PMID 10360331. Bibcode 1998MSRv...17..369M.
- ↑ Analysis of phosphorylation sites on proteins from Saccharomyces cerevisiae by electron transfer dissociation (ETD) mass spectrometry. Proceedings of the National Academy of Sciences of the United States of America. 2007, s. 2193–2198. DOI 10.1073/pnas.0607084104. PMID 17287358. Bibcode 2007PNAS..104.2193C.
- ↑ C. K. Frese; A. F. Altelaar; H. van den Toorn; D. Nolting; J. Griep-Raming; A. J. Heck; S. Mohammed. Toward full peptide sequence coverage by dual fragmentation combining electron-transfer and higher-energy collision dissociation tandem mass spectrometry. Analytical Chemistry. 2012, s. 9668–9673. DOI 10.1021/ac3025366. PMID 23106539.
- ↑ C. K. Frese; H. Zhou; T. Taus; K. Mechtler; A. J. Heck; S. Mohammed. Unambiguous phosphosite localization using electron-transfer/higher-energy collision dissociation (EThcD). Journal of Proteome Research. 2013, s. 1520–1525. DOI 10.1021/pr301130k. PMID 23347405.
- ↑ J. J. Coon; J. Shabanowitz; D. F. Hunt; J. E. Syka. Electron transfer dissociation of peptide anions. Journal of the American Society for Mass Spectrometry. 2005, s. 880–882. DOI 10.1016/j.jasms.2005.01.015. PMID 15907703.
- ↑ B. A. Budnik; K. F. Haselmann; R. A. Zubarev. Electron detachment dissociation of peptide di-anions: an electron–hole recombination phenomenon. Chemical Physics Letters. 2001, s. 299–302. DOI 10.1016/S0009-2614(01)00501-2. Bibcode 2001CPL...342..299B.
- ↑ K. Chingin; A. Makarov; E. Denisov; O. Rebrov; R. A. Zubarev. Charge transfer dissociation (CTD) mass spectrometry of peptide cations using kiloelectronvolt helium cations. Analytical Chemistry. 2014, s. 372–379. DOI 10.1021/ac403193k. PMID 24236851.
- ↑ W. D. Hoffmann; G. P. Jackson. Fragmentation of positively-charged biological ions activated with a beam of high-energy cations. Journal of the American Society for Mass Spectrometry. 2014, s. 1939–1943. DOI 10.1007/s13361-014-0989-6. PMID 25231159. Bibcode 2014JASMS..25.1939H.
- ↑ J. W. Morgan; J. M. Hettick; D. H. Russell. Peptide sequencing by MALDI 193-nm photodissociation TOF MS. [s.l.]: [s.n.], 2005. Dostupné online. ISBN 9780121828073. DOI 10.1016/S0076-6879(05)02006-9. PMID 16401510. Kapitola Peptide Sequencing by MALDI 193‐nm Photodissociation TOF MS, s. 186–209.
- ↑ D. P. Little; J. P. Speir; M. W. Senko; P. B. O'Connor; F. W. McLafferty. Infrared multiphoton dissociation of large multiply charged ions for biomolecule sequencing. Analytical Chemistry. 1994, s. 2809–2815. DOI 10.1021/ac00090a004. PMID 7526742.
- ↑ a b P. D. Schnier; W. D. Price; R. A. Jockusch; E. R. Williams. Blackbody infrared radiative dissociation of bradykinin and its analogues: energetics, dynamics, and evidence for salt-bridge structures in the gas phase. Journal of the American Chemical Society. 1996, s. 7178–4189. DOI 10.1021/ja9609157. PMID 16525512.
- ↑ R. C. Dunbar. BIRD (blackbody infrared radiative dissociation): evolution, principles, and applications. Mass Spectrometry Reviews. 2004, s. 127–158. DOI 10.1002/mas.10074. PMID 2004MSRv...23..127D.
- ↑ Verena Grill; Jianwei Shen; Chris Evans; R. Graham Cooks. Collisions of ions with surfaces at chemically relevant energies: Instrumentation and phenomena. Review of Scientific Instruments. 2001, s. 3149. DOI 10.1063/1.1382641. Bibcode 2001RScI...72.3149G.
- ↑ M. Mabud. Surface-induced dissociation of molecular ions. International Journal of Mass Spectrometry and Ion Processes. 1985, s. 285–294. DOI 10.1016/0168-1176(85)83024-X. Bibcode 1985IJMSI..67..285M.
- ↑ a b Alyssa Stiving; Zachary VanAernum; Florian Busch; Sophie Harvey; Samantha Sarni; Vicki Wysocki. Surface-Induced Dissociation: An Effective Method for Characterization of Protein Quaternary Structure. Analytical Chemistry. 2018-11-09, s. 190–191. DOI 10.1021/acs.analchem.8b05071. PMID 30412666.
- ↑ Royston S. Quintyn; Mowei Zhou; Jing Yan; Vicki H. Wysocki. Surface-Induced Dissociation Mass Spectra as a Tool for Distinguishing Different Structural Forms of Gas-Phase Multimeric Protein Complexes. Analytical Chemistry. 2015-12-01, s. 11 879 – 11 886. ISSN 0003-2700. DOI 10.1021/acs.analchem.5b03441. PMID 26499904.
- ↑ Julia Laskin; Jean H. Futrell. Activation of large lons in FT-ICR mass spectrometry. Mass Spectrometry Reviews. 2005, s. 135–167. Dostupné online. ISSN 0277-7037. DOI 10.1002/mas.20012. PMID 15389858. Bibcode 2005MSRv...24..135L.
- ↑ Electron transfer dissociation in the hexapole collision cell of a hybrid quadrupole-hexapole Fourier transform ion cyclotron resonance mass spectrometer. Rapid Communications in Mass Spectrometry. 2008, s. 271–278. ISSN 0951-4198. DOI 10.1002/rcm.3356. PMID 18181247. Bibcode 2008RCMS...22..271KL.
- ↑ Julia Laskin. Surface-Induced Dissociation: A Unique Tool for Studying Energetics and Kinetics of the Gas-Phase Fragmentation of Large Ions. European Journal of Mass Spectrometry. 2015, s. 377–389. ISSN 1469-0667. DOI 10.1255/ejms.1358. PMID 26307719.
- ↑ S. E. Ong; M. Mann. Mass spectrometry-based proteomics turns quantitative. Nature Chemical Biology. 2005, s. 252–262. DOI 10.1038/nchembio736. PMID 16408053.
- ↑ M. Bantscheff; M. Schirle; G. Sweetman; J. Rick; B. Kuster. Quantitative mass spectrometry in proteomics: a critical review. Analytical and Bioanalytical Chemistry. 2007, s. 1017–1031. DOI 10.1007/s00216-007-1486-6. PMID 17668192.
- ↑ M. Nikolov; C. Schmidt; H. Urlaub. Quantitative Methods in Proteomics. [s.l.]: [s.n.], 2012. ISBN 978-1-61779-884-9. DOI 10.1007/978-1-61779-885-6_7. PMID 22665296. Kapitola Quantitative mass spectrometry-based proteomics: an overview, s. 85–100.
- ↑ S. Maher; F. P. Jjunju; S. Taylor. 100 years of mass spectrometry: Perspectives and future trends. Reviews of Modern Physics. 2015, s. 113–135. DOI 10.1103/RevModPhys.87.113. Bibcode 2015RvMP...87..113M.
- ↑ Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents. Molecular & Cellular Proteomics. 2004, s. 1154–1169. DOI 10.1074/mcp.M400129-MCP200. PMID 15385600.
- ↑ L. R. Zieske. A perspective on the use of iTRAQ reagent technology for protein complex and profiling studies. Journal of Experimental Botany. 2006, s. 1501–1508. DOI 10.1093/jxb/erj168. PMID 16574745.
- ↑ P. R. Gafken; P. D. Lampe. Methodologies for characterizing phosphoproteins by mass spectrometry. Cell Communication & Adhesion. 2006, s. 249–262. DOI 10.1080/15419060601077917. PMID 17162667.
- ↑ Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 2003, s. 1895–1904. DOI 10.1021/ac0262560. PMID 12713048.
- ↑ T. E. Angel; U. K. Aryal; S. M. Hengel; E. S. Baker; R. T. Kelly; E. W. Robinson; R. D. Smith. Mass spectrometry-based proteomics: existing capabilities and future directions. Chemical Society Reviews. 2012, s. 3912–3928. DOI 10.1039/c2cs15331a. PMID 22498958.
- ↑ J. W. Robinson. Protein sequence information by matrix-assisted laser desorption/ionization in-source decay mass spectrometry. Mass Spectrometry Reviews. 2007, s. 672–682. DOI 10.1002/mas.20142. PMID 17492750. Bibcode 2007MSRv...26..672H.
- ↑ I. Shadforth; D. Crowther; C. Bessant. Protein and peptide identification algorithms using MS for use in high-throughput, automated pipelines. Proteomics. 2005, s. 4082–4095. DOI 10.1002/pmic.200402091. PMID 16196103.
- ↑ E. Mørtz; P. B. O'Connor; P. Roepstorff; N. L. Kelleher; T. D. Wood; F. W. McLafferty; M. Mann. Sequence tag identification of intact proteins by matching tanden mass spectral data against sequence data bases. Proceedings of the National Academy of Sciences of the United States of America. 1996, s. 8264–8267. DOI 10.1073/pnas.93.16.8264. PMID 8710858. Bibcode 1996PNAS...93.8264M.
- ↑ P. Roepstorff; J. Fohlman. Proposal for a common nomenclature for sequence ions in mass spectra of peptides. Biomedical Mass Spectrometry. 1984, s. 601. DOI 10.1002/bms.1200111109. PMID 6525415.
- ↑ Richard S. Johnson; Stephen A. Martin; Klaus Biemann. Collision-induced fragmentation of (M + H)+ ions of peptides. Side chain specific sequence ions. International Journal of Mass Spectrometry and Ion Processes. 1988, s. 137–154. DOI 10.1016/0168-1176(88)80060-0. Bibcode 1988IJMSI..86..137J.
- ↑ A. M. Falick; W. M. Hines; K. F. Medzihradszky; M. A. Baldwin; B. W. Gibson. Low-mass ions produced from peptides by high-energy collision-induced dissociation in tandem mass spectrometry. Journal of the American Society for Mass Spectrometry. 1993, s. 882–893. DOI 10.1016/1044-0305(93)87006-X. PMID 24227532.
- ↑ Kevin M. Downard; Klaus Biemann. Methionine specific sequence ions formed by the dissociation of protonated peptides at high collision energies. Journal of Mass Spectrometry. 1995, s. 25–32. DOI 10.1002/jms.1190300106. Bibcode 1995JMSp...30...25D.
- ↑ Kevin M. Downard; Klaus Biemann. Mass spectrometry of oligosaccharides. Mass Spectrometry Reviews. 2004, s. 161–227. Dostupné online. DOI 10.1002/mas.10073. PMID 14966796. Bibcode 2004MSRv...23..161Z.
- ↑ Bruno Domon; Catherine E. Costello. A systematic nomenclature for carbohydrate fragmentations in FAB-MS/MS spectra of glycoconjugates. Glycoconj. J.. 1988, s. 397–409. DOI 10.1007/BF01049915.
- ↑ E. Spina; R. Cozzolino; E. Ryan; D. Garozzo. Sequencing of oligosaccharides by collision-induced dissociation matrix-assisted laser desorption/ionization mass spectrometry. Journal of Mass Spectrometry. 2000, s. 1042–1048. DOI 10.1002/1096-9888(200008)35:8<1042::AID-JMS33>3.0.CO;2-Y. PMID 10973004. Bibcode 2000JMSp...35.1042S.
- ↑ J. H. Banoub; R. P. Newton; E. Esmans; D. F. Ewing; G. Mackenzie. Recent developments in mass spectrometry for the characterization of nucleosides, nucleotides, oligonucleotides, and nucleic acids. Chemical Reviews. 2005, s. 1869–1915. DOI 10.1021/cr030040w. PMID 15884792.
- ↑ B. Thomas; A. V. Akoulitchev. Mass spectrometry of RNA. Trends in Biochemical Sciences. 2006, s. 173–181. DOI 10.1016/j.tibs.2006.01.004. PMID 16483781.
- ↑ J. Wu; S. A. McLuckey. Gas-phase fragmentation of oligonucleotide ions. International Journal of Mass Spectrometry. 2004, s. 197–241. DOI 10.1016/j.ijms.2004.06.014. Bibcode 2004IJMSp.237..197W.
- ↑ Thomas E. Angel; Uma K. Aryal; Shawna M. Hengel; Erin S. Baker; Ryan T. Kelly; Errol W. Robinson; Richard D. Smith. Mass spectrometry based proteomics: existing capabilities and future directions. Chemical Society Reviews. 2012-05-21, s. 3912–3928. ISSN 0306-0012. DOI 10.1039/c2cs15331a. PMID 22498958.
- ↑ Xuemei Han; Aaron Aslanian; John R. Yates. Mass Spectrometry for Proteomics. Current Opinion in Chemical Biology. 2008, s. 483–490. ISSN 1367-5931. DOI 10.1016/j.cbpa.2008.07.024. PMID 18718552.
Média použitá na této stránce
Autor: Michael Pereckas, Licence: CC BY-SA 2.0
With the top cover removed, the ion trap itself and the detector dynodes can be seen in this shot from the front of the instrument.
Autor: K. Murray (Kkmurray), Licence: CC BY-SA 3.0
Schematic representation of a tandem mass spectrometry experiment
Autor: Florian H. Tirk (Tirkfl), Licence: CC BY-SA 3.0
Example of a full MS spectrum and an MS/MS spectrum of a peptide.
Autor: Egor Vorontsov, Licence: CC BY 4.0
Isobaric labeling mass spectrometry-based proteomic workflow with 4 unique reagents in a set, and 7 different biological samples combined into 2 labeling groups (plexes). The shape and exaggerated width of chromatographic peaks do not represent experimental measurement.